GEORGIKON MEZŐGAZDASÁGTUDOMÁNYI KAR Állattudományi és Állattenyésztéstani Tanszék
Tanszékvezető Dr. Husvéth Ferenc egyetemi tanár, az MTA doktora DOKTORI (PhD) ÉRTEKEZÉS
Készült a Pannon Egyetem Állattenyésztési Tudományok Doktori Iskola, jogutód: Állat- és Agrárkörnyezet-tudományi Doktori Iskola
állattenyésztés tudományok tudományág keretében
Doktori Iskola volt vezetője Doktori Iskola jelenlegi vezetője Dr. Szabó Ferenc Dr. Anda Angéla
egyetemi tanár, az MTA doktora egyetemi tanár, az MTA doktora
Témavezető Társ-témavezető
Dr. Husvéth Ferenc Dr. Fébel Hedvig egyetemi tanár, az MTA doktora tudományos tanácsadó
Konzulens Bán Beáta vizsgáló mérnök
DNS MIKROSZATELLITEK VIZSGÁLATA LÓ ÉS SZARVASMARHA FAJTÁKBAN
Készítette Józsa Csilla KESZTHELY
2008
DNS MIKROSZATELLITEK VIZSGÁLATA LÓ ÉS SZARVASMARHA FAJTÁKBAN
Értekezés doktori (PhD) fokozat elnyerése érdekében Írta:
JÓZSA CSILLA
Készült a Pannon Egyetem Állattenyésztési Tudományok Doktori Iskola, jogutód: Állat- és Agrárkörnyezet-tudományi Doktori Iskola
állattenyésztés tudományok tudományág keretében Témavezető: Dr.Husvéth Ferenc, egyetemi tanár, az MTA doktora Elfogadásra javaslom (igen / nem) ……...
A jelölt a doktori szigorlaton …... % -ot ért el.
Keszthely, 2007.
Az értekezést bírálóként elfogadásra javaslom:
Bíráló neve: …...…... igen /nem ……….
Bíráló neve: …... igen / nem ……….
A jelölt az értekezés nyilvános vitáján …...% - ot ért el.
Keszthely, 2008. ……….
a Bíráló Bizottság elnöke A doktori (PhD) oklevél minősítése…...
………
Az EDT elnöke
TARTALOMJEGYZÉK oldal
1. BEVEZETÉS ...7
2. CÉLKITŰZÉSEK ...9
3. IRODALMI ÁTTEKINTÉS ... 11
3.1. A molekuláris genetika alapjai ... 11
3.1.1. Az örökítő anyag, a DNS molekula ... 11
3.1.2. Mikroszatellit (STR) vizsgálatok ... 13
3.1.3. PCR – polimeráz láncreakció (polymerase chain reaction) ... 16
3.1.3.1. A PCR sikertörténete ... 16
3.1.3.2. A polimeráz láncreakció elve ... 16
3.1.3.3. A PCR reakció menete ... 17
3.1.3.4. A PCR reakcióelegy összetevői ... 19
3.1.4. A kapilláris gélelektroforézis ... 20
3.2. Molekuláris genetika az állattenyésztésben... 23
3.2.1. Genetikai markerek alkalmazása az állattenyésztésben ... 24
3.2.2. Molekuláris genetika a génmegőrzésben ... 25
3.2.3. Molekuláris genetika a származásellenőrzésben ... 27
3.2.3.1. A DNS–re alapozott származásellenőrzés módszerének főbb vonatkozásai ... 28
3.2.3.2. Származásellenőrzés Magyarországon ... 29
3.2.3.2.1. Származásellenőrzés a lótenyésztésben ... 29
3.2.3.2.1. Származásellenőrzés a szarvasmarha tenyésztésben ... 30
4. ANYAG ÉS MÓDSZER ... 31
4.1. A kísérleti minták gyűjtése és előkészítése a molekuláris genetikai vizsgálatokhoz ... 31
4.1.1. A vizsgálatokba vont fajták, populációk ... 34
4.1.1.1. Ló faj ... 34
4.1.1.2. Szarvasmarha faj ... 34
4.1.2. A DNS kivonása és tisztítása ... 36
4.2. A molekuláris genetikai elemzések során alkalmazott laboratóriumi vizsgálatok ... 37
4.2.1. STR lókuszok vizsgálatának módszere ló faj esetén ... 37
4.2.1.1. A vizsgálandó szakaszok amplifikálása ... 37
4.2.1.2. Az STR fragmensek genotipizálása (elválasztás, detektálás, szelektálás) ... 38
4.2.2. STR lókuszok vizsgálatának módszere szarvasmarha faj esetén .. 40
4.2.2.1. A vizsgálandó szakaszok amplifikálása ... 40
4.2.2.2. Az STR fragmensek genotipizálása (elválasztás, detektálás, szelektálás) ... 41
4.3. Kísérleti eredmények értékelése, statisztikai analízisek ... 44
5. EREDMÉNYEK ... 47
5.1. A vizsgált fajták genetikai változatosságának leírása DNS mikroszatellit markerek segítségével ... 47
5.1.1. A vizsgált ló fajták genetikai változatosságának jellemzése ... 47
5.1.1.1. Allélfrekvencia értékek; allélszámok (megfigyelt, effektív); Shanonn-féle információs index; egyedi, fajtaspecifikus allélek; allélgazdagság (Ar) ... 47
5.1.1.2. Várt és megfigyelt heterozigozitás (He, Ho), Nei-féle várt heterozigozitás (Hexp) ... 55
5.1.1.3. Hardy-Weinberg egyensúlytól való eltérés (PH-W), Wright-féle
fixációs index (FIS) ... 57
5.1.1.4. Nei-féle genetikai távolság mérőszámai és a távolságok dendogrammon való ábrázolása ... 59
5.1.1.5. Fajtapárok közötti fixációs index (FST) és génáramlás (Nm) ... 60
5.1.1.6. Származásellenőrzés DNS mikroszatellitek segítségével ... 60
5.1.2. A vizsgált húsmarha fajták genetikai változatosságának jellemzése ... 63
5.1.2.1. Allélfrekvencia értékek; allélszámok (megfigyelt, effektív); Shanonn-féle információs index; egyedi, fajtaspecifikus allélek; allélgazdagság (Ar) ... 63
5.1.2.2. Várt és megfigyelt heterozigozitás (He, Ho), Nei-féle várt heterozigozitás (Hexp) ... 70
5.1.2.3. Hardy-Weinberg egyensúlytól való eltérés (PH-W), Wright-féle fixációs index (FIS) ... 72
5.1.2.4. Nei-féle genetikai távolság mérőszámai és a távolságok dendogrammon való ábrázolása ... 75
5.1.2.5. Fajtapárok közötti fixációs index (FST) és génáramlás (Nm) ... 76
5.1.2.6. Származásellenőrzés DNS mikroszatellitek segítségével ... 76
5.2. Kiválasztott populációk genetikai jellemzése, összehasonlítása DNS markerek segítségével ... 79
5.2.1. A dióspusztai és a kerteskői angol telivér populációk genetikai összehasonlítása ... 79
5.2.2. A derecskei és a hajdúböszörményi magyar tarka populációk genetikai összehasonlítása ... 83
6. EREDMÉNYEK ÉRTÉKELÉSE ... 88
6.1. DNS lókuszok vizsgálata során kapott eredmények értékelése ló fajban... 88
6.2. DNS lókuszok vizsgálata során kapott eredmények értékelése szarvasmarha fajban... 92
7. KÖVETKEZTETÉSEK ... 96
8. ÚJ EREDMÉNYEK ... 98
9. ÖSSZEFOGLALÁS ... 100
10. PUBLIKÁCIÓK JEGYZÉKE ... 103
11. IRODALOMJEGYZÉK ... 107
TÁBLÁZATOK JEGYZÉKE ... 124
ÁBRÁK JEGYZÉKE ... 126
KÖSZÖNETNYILVÁNÍTÁS... 128
MELLÉKLETEK ... 129
KIVONAT
DNS MIKROSZATELLITEK VIZSGÁLATA LÓ ÉS SZARVASMARHA FAJTÁKBAN
A szerző a doktori munkája során néhány hazánkban is tenyésztett ló- (angol telivér, amerikai ügető, lipicai, magyar hidegvérű) és szarvasmarhafajta (magyar tarka, hereford, limousin, charolais, aberdeen angus) genetikai jellemzőit vizsgálta DNS mikroszatellitek segítségével, valamint ezen markerek alkalmazhatóságát becsülte a hazai állattenyésztés fejlesztése érdekében.
Az vizsgálatok folyamán a ló fajban 17 (VHL20, HTG4, AHT4, HMS7, HTG6, AHT5, HMS6, ASB23, ASB2, HTG10, HTG7, HMS3, HMS2, ASB17, LEX3, HMS1, CA425), a szarvasmarha fajban 11 (BM1824, BM2113, ETH10, ETH225, INRA23, SPS115, TGLA122, TGLA126, TGLA227, ETH3, TGLA53) mikroszatellit detektálására került sor.
Az eredmények alapján a több genetikai markerre alapozott analitikai eljárások alkalmasnak mutatkoztak az egyedek és a fajták közötti különbségek, a fajták genetikai változatosságának meghatározására. Az eredmények bizonyították, hogy a DNS mikroszatellit vizsgálatok szembetűnő különbségeket jeleztek a vizsgált ló- és szarvasmarha fajták között, s megállapítható, hogy a hazai állományok egésze genetikailag meglehetősen heterogén volt és jelentős genetikai erőforrásokkal rendelkezett. Mindezek mellett a disszertációban tanulmányozott fajták a beltenyésztettség jeleit mutatták, amit több genetikai mérőszámmal (várt és megfigyelt heterozigozitás egymáshoz viszonyítása; FIS értékek pozitivitása, illetve negativitása) is igazolt a szerző. Az egyedek származása az alkalmazott biotechnológiai módszer segítségével egyértelműen eldönthető volt (a vizsgált szarvasmarha fajták esetében 97,85 és 99,46%-os, a vizsgált lófajták esetében 99,80 és 99,98%-os biztonsággal), így megbízhatóan alkalmazható a hazai rutin származásellenőrzésben is.
ABSTRACT
DNA MICROSATELLITE ANALYSIS OF HORSE AND CATTLE BREEDS
In this doctoral work the author investigated the genetic characteristics of several horse and cattle breeds in Hungary with DNA microsatellites.
Moreover, the author estimated the applicability of these markers in order to improve animal husbandry in the country.
Seventeen horse and eleven cattle microsatellites were detected in breeds throughout the study.
According to the results, the applied analysis methods based on several genetic markers were suitable for identifying differences between individual animals and breeds and to determine the genetic variability of breeds. DNA microsatellite analyses revealed significant differences between the investigated horse and cattle breeds. These findings suggest that horse and cattle breeds in Hungary are genetically considerably heterogeneous and that they have significant genetic resources. Nevertheless, breeds studied in present
thesis showed signs of inbreeding, which was confirmed by various genetic parameters (comparison of the expected and observed heterozygosity, positivity and negativity of FIS values). The biotechnological method applied could unambiguously determine the origin of the individual animals (97.85 - 99.46% and 99.80 – 99.98% confidence for the examined cattle and horse breeds respectively). Thus, this method can be reliably used in routine parentage control in Hungary as well.
AUSZUG
UNTERSUCHUNG DER MIKROSATELITEN IN PFERDE- UND RINDSORTEN
Der Verfasser untersuchte während seiner Doktorarbeit die genetische Charakteristika einiger in Ungarn auch gezüchteten Pferde- und Rindsorten mit Hilfe der DNS-Mikrosateliten, sowie geschätzte die Anwendbarkeit dieser Marker im Interesse der Entwicklung der ungarischen Viehzucht.
In den untersuchungen wurden bei den Pferden 17, bei den Rinden 11 Mikrosateliten detektiert.
Die auf mehreren genetischen Markern basierende analytische Verfahren erwiesen sich anhand unserer Ergebnisse geeignet für die Bestimmung der Unterschiede zwischen den einzelnen Tieren und zwischen den verschiedenen Sorten, sowie auch für die Bestimmung der genetischen Vielfalt der Sorten. Die DNS-Mikrosateliten-Untersuchungen zeigten hervorstechende Unterschiede zwischen den untersuchten Pferd- und Rindsorten, und es kann auch festgestellt werden, dass die ganze ungarische Population genetisch ziemlich heterogen war, und über bedeutende genetische Ressourcen verfügte. Daneben zeigten die in der Dissertation untersuchte Sorten die Anzeichen der Innzucht, die vom Verfasser auch mit mehreren genetischen Messzahlen (Vergleich der erwarteten und beobachteten Heterozigosität; Positivität bzw. Negativität der FIS Werte) bestätigt wurde. Die Herkunft der einzelnen Tiere konnte mit Hilfe der angewendeten biotechnologischen Methode eindeutig entschieden werden (bei den untersuchten Rindsorten mit einer Sicherheit von 97,85 und 99,46%, bei den Pferdesorten von 99,80 und 99,98%), so kann diese Methode in der routinemäßig erfolgenden Herkunftskontrolle verlässlich angewendet werden.
…Lehet, hogy nem a biokémikus fogja kimondani az utolsó szót az élet leírásában. Az azonban biztos, hogy ezt az utolsó szót nélküle nem fogják kimondani…
Sir Frederick Gowland Hopkins
1. BEVEZETÉS
A molekuláris genetikai kutatások számos új felfedezéssel gazdagodtak az elmúlt évtizedben. Napról napra újabb információkat publikáltak a kutatók, amelyek elősegítették a molekuláris biológia fejlődését. Ezek a kutatások nagymértékben befolyásolták az élelmiszertermelésben, ezen belül az állattenyésztés területén bekövetkezett változásokat. A több és jobb minőségű állati eredetű élelmiszer előállítását nem az állatlétszám növelésével, hanem elsősorban a termelés hatékonyságának fokozásával kell megoldani. Eközben természetesen az állatvédelemre, környezetvédelemre és a biodiverzitás megőrzésére irányuló törekvéseknek és az ezekre vonatkozó törvényeknek is eleget kell tennünk. Az állattenyésztés csak úgy tud megfelelni a fokozott mennyiségi és minőségi elvárásoknak, ha új és jobb módszereket alkalmaz a tenyésztés, a takarmányozás, a management, az állategészségügy, az állatvédelem, a génmegőrzés, a termék-feldolgozás, a termékértékesítés és élelmiszerbiztonság területén (Fésüs és mtsai, 2000). Ezért nagyon fontos, hogy a hazai kutatómunka a napjainkban zajló genetikai és biotechnológiai forradalom új felfedezéseiből született eljárásokat, módszereket alkalmazza és felhasználja az állattenyésztés, az állatnemesítés területén is.
A szelekció segítségével az ember a háziasítás kezdete óta beleszólt az általa tenyésztett populáció genetikai szerkezetébe. Ezen munka eredményeként alakult ki a ma élő tenyésztett fajok ismert tulajdonságainak változatossága. A hagyományos szelekciós módszerek mellett azonban folyamatosan megjelennek a molekuláris genetika által teremtett új eljárások, lehetőségek, amelyek megbízhatóbb eredményeket nyújtanak mind a kutatók, mind a tenyésztők számára. Napjainkig még remény sem volt arra, hogy a QTL-eknél (Quantitative Trait Loci = mennyiségi jellegekért felelős lókuszok) egyszerűbb öröklésmenetet mutató termelési tulajdonságok molekuláris
genetikai hátterét fel lehessen tárni (Fésüs és mtsai, 2000). A genetika fejlődése mostanra elérte azt a szintet, hogy már néhány fenotípus genetikai meghatározottsága ismert, s ez a kör még bővülni fog. Ezáltal a genetikai vizsgálatokkal közvetlenül a DNS szekvencia alapján megállapítható az, hogy egy egyed hordoz-e egy adott tulajdonságot, vagy sem.
A sok megoldásra váró feladat ellenére nem lehet kétségbe vonni azt, hogy a molekuláris biotechnológia, a DNS-technológia és az egyre tökéletesedő géntérképek döntő módon hozzájárulnak a genetikai és fiziológiai ismereteink bővítéséhez, és mindez az állattenyésztés előrehaladásának, fejlődésének alapját képezi. Emellett a rohamosan fejlődő komputertechnika, a közvetlen és az azonnali kapcsolatfelvétel a kutatók között egyre inkább lerövidíti a molekuláris genetika és immunológia eredményeinek átviteli idejét a tenyésztési gyakorlatba, az ott történő ellenőrzésüket, nemkülönben a genetikai markerek által segített szelekció elterjedését.
2. CÉLKITŰZÉSEK
A Mezőgazdasági Szakigazgatási Hivatal (MgSzH), Állattenyésztési Igazgatóság, Központi Állattenyésztési Laboratóriuma (Budapest) több mint 15 éve végez vércsoport és biokémiai polimorfizmus vizsgálatokat származásellenőrzés céljából, de az elmúlt években fokozatosan áttértek a DNS mikroszatellitek alkalmazására azok megbízhatósága és nagyszámú polimorfizmusuk miatt. Az eddigi vizsgálatok azt mutatjáják, hogy a DNS mikroszatellitek rendkívül variabilis lókuszok, és ezért segítségükkel a populációk genetikai variabilitásának finom struktúrája pontosan feltárható, valamint egyedek származása is könnyen igazolható. Bár a módszer hazánkban még viszonylag újnak számít, a jelenleg ismert DNS-technikák közül a mikroszatellitek vizsgálata egyértelműen hasznosnak ígérkezik a populációszintű variabilitás tanulmányozására a hazánkban tenyésztett gazdasági állatfajok tekintetében is.
A disszertáció tárgyát képező vizsgálatok főbb célkitűzései az alábbiakban foglalhatók össze:
A Magyarországon tenyésztett ló (angol telivér, amerikai ügető, lipicai, magyar hidegvérű), illetve szarvasmarha (magyar tarka, limousin, hereford, charolais, aberdeen angus) fajták genetikai változatosságának feltérképezése DNS mikroszatellit markerek segítségével.
A mikroszatellit lókuszokon az allélok számának és frekvenciájának meghatározása, egyedi allélek keresése az előzőekben leírt ló, illetve szarvasmarha fajtákban.
A hazánkban tenyésztett egyes ló és szarvasmarha fajták közötti genetikai távolság felmérése DNS mikroszatellit vizsgálatok segítségével.
A beltenyésztettség mértékének felderítése DNS mikroszatellit vizsgálatok alapján a kísérletekbe vont populációkban, illetve fajtákban.
A DNS mikroszatellit markerek alkalmazhatóságának becslése a hazai állattenyésztés fejlesztése érdekében.
3. IRODALMI ÁTTEKINTÉS
3.1. A molekuláris genetika alapjai 3.1.1. Az örökítő anyag, a DNS molekula
Friedrich Miescher (1869; 1871) izolált biológiai anyagból svájci laboratóriumában először nukleinsavat. Oswald Avery, Colin MacLeod és Maclyn McCarty (Rockefeller Intézet, New York) 1944-ben írták le először gondosan végzett, hosszadalmas baktériumkísérletek nyomán, hogy a DNS az öröklődés anyaga (Avery és mtsai, 1944). Felfedezésüket vagy közömbösség, vagy pedig hitetlenség fogadta, és a tudóscsoport vezetőjének, Averynek a visszahúzódó személyisége nem engedte meg a felfedezés propagálását. Ha kevesen is, de voltak azonban, akik felismerték a megállapítás forradalmi jellegét. Így Erwin Chargaff, a Columbia Egyetem akkor már híres biokémikusa a közlemény hatására egyik pillanatról a másikra abbahagyta addigi munkáit és további munkásságát a DNS biokémiájának szentelte.
Hamarosan megállapította, hogy a DNS nem olyan monoton felépítésű, mint amilyennek feltételezték. Ellenkezőleg, összetétele a biológiai forrás függvénye: a különböző élőlényekben a DNS-ben található bázisok aránya az adott élőlényre specifikus, az (A: adenin + T: timin)/(G: guanin + C: citozin) arány széles határok között változhat (Hargittai, 2003). Chargaff másik fontos felfedezése az volt, hogy minden szervezet DNS-molekulájában guaninból és citozinból azonos mennyiség van jelen, és ugyanígy azonos mennyiségben van jelen adenin és timin, vagyis a G/C és az A/T arányok értékei egynek felelnek meg. Ez utóbbi megállapítás volt később az alapja a bázispárok feltételezésének a kettőscsavar-szerkezetben Watson és Crick szerint (Venetianer, 1998). Miután 1952-ben Chargaff igazolta a DNS molekulát alkotó négy szerves bázis párosodás szabályát, 1953-ban James Watson és Francis Crick közzé tette azt a korszakalkotó dolgozatot a Nature című tudományos folyóiratban, amelyben leírják a DNS szerkezetét, a double-helix (kettős spirál) szerkezetet (Watson és Crick, 1953a, 1953b). A vizsgálatok középpontjában tehát a DNS-molekula állt, amely az örökletes tulajdonságok hordozója. A DNS dezoxiribonukleotidokból lineárisan felépített polimer molekula, amely minden élő sejtben előfordul (Weaver és Hedrick, 2000).
1. ábra: A DNS molekula
(forrás: www.enc.hu/1enciklopedia/fogalmi/biol_mol/dns.htm)
A genetikai információt az eukariotákban a DNS molekula bázissorrendje hordozza. A DNS rendszerint két egymást kiegészítő (komplementer) láncból áll, amelyek egy jobbra csavarodó spirált alkotnak. A DNS molekula gerincét általában két párhuzamosan futó, egymás körül felcsavarodott cukorfoszfát lánc képezi (1. ábra). A DNS lánc cukor és foszfát egységeit aszimmetrikus 5'- 3' foszfodiészter kötések kapcsolják össze. Ennek megfelelően a DNS szál is polarizált: egyik végén a terminális nukleotid 5' szénatomján foszforil gyök, a másik végén a terminális nukleotid 3' szénatomján hidroxil gyök van. A két párhuzamos, de ellenkező irányban futó cukorfoszfát gerincen a cukorkomponensekhez négyféle bázis (A,T,G,C) kapcsolódik. A bázisok a kiegészítő lánc ellentett bázisai felé mutatnak, és az ellentett bázisokhoz hidrogénkötésekkel kapcsolódnak. A bázisok szerkezetéből adódóan az egyik láncon lévő A (adenin) a másik lánc T (timin)-jével két hidrogén kötéssel, a G
(guanin) a C (citozin)-nal három hidrogén kötéssel kapcsolódik (Weaver és Hedrick, 2000).
Az egyedekre jellemző tulajdonságok a DNS-ben rögzültek, kódoltak. A DNS-nek vannak olyan szakaszai, melyek egy-egy tulajdonság kialakításáért felelősek (gének), más régiók pedig a gének működését szabályozzák (Khorana, 1968). Vannak azonban olyan DNS darabok, melyek az előbb említett genetikai információt nem hordoznak, hanem hosszabb-rövidebb ismétlődő szekvenciák (Fésüs és mtsai, 1997). Ezek a repetitív szakaszok az egész genom területén elszórtan megtalálhatók, néha akár a DNS több mint 30
%-át is kitehetik. Bizonyos kromoszóma-szegmentumokban – mint például a centromér régiókban – minden ismétlődő szekvencia, de különösen a tetra- nukleotidok ismétlődésének a gyakorisága magasabb. A repetitív DNS biológiai funkciójáról meglehetősen hiányosak az ismereteink, de a legtöbb eukariótánál azonos alapelvet felmutató szerveződése arra utal, hogy ez a frakció fontos strukturális és funkcionális szereppel bír. A repetitív DNS szerkezetében gyakran a közeli rokonságban álló genotípusok között is nagy mértékű változatosságot tapasztalhatunk, így a repetitív szekvenciákon alapuló markerek információs értéke magas. A repetitív DNS az ismétlődő egységek hossza alapján felosztható miniszatellitekre és mikroszatellitekre (Fésüs és mtsai, 1997).
3.1.2. Mikroszatellit (STR) vizsgálatok
A mikroszatellitek abba a repetitív szekvencia családba tartoznak, amelyben igen egyszerű, néhány bázis ill. szekvencia ismétlődik egy szakaszon, ugyanakkor a kópiaszám nagy mértékű variabilitást mutat. A tandem ismétlődő régiókat VNTR-nek (Variable Number Tandem Repeats = változó számú tandem ismétlődések, Nakamura és mtsai 1987), STR-nek (Short Tandem Repeat = rövid tandem ismétlődés, Edwards és mtsai, 1991) mikroszatellitnek (Litt és Luty, 1989) vagy SSR-nek (Simple Sequence Repeat
= egyszerű szekvencia ismétlődés, Jacob és mtsai, 1991) nevezzük. A mikroszatellitek 2-5 bp (bázispár) hosszú, könnyen amplifikálható DNS szakaszok, amelyek egyenletesebben oszlanak el a genomban, mint a miniszatellitek (Buduram és mtsai, 2005; Litt és Luty, 1989; Weber és May,
1989; Tautz és Renz, 1984; Jeffreys és mtsai, 1985). Az alapmotívumok száma alapján megkülönböztetünk di-, tri-, tetra- és pentanukleotidokat. A mikroszatellitek nagy polimorfizmust mutatnak, ami azt jelenti, hogy egy populációban, meghatározott génhelyen (lókusz) allélok egész sora azonosítható (De Woody és Avise, 2000). A különböző allélok a megismételt egységek eltérő számából jönnek létre (pl. (CA)15, (CA)17). A repetitív minta kedvez a mutációnak, ami az egyik oka a mikroszatellitek nagy polimorfizmusának. A mikroszatellitek mutációs rátáját 10-4 és 5x10-6-ra becsülik (Edwards és mtsai, 1992; Dallas, 1992; Craighead és mtsai, 1995). A mutáció mechanizmusa régóta vitatott (Levinson és Gutman, 1987; Richards és Sutherland, 1994), csak a különböző hosszvarianciákat eredményező fő mechanizmus a biztos, amely a DNS replikáció során bekövetkező hiba eredménye.
A mikroszatellitek kiváló eszközei a géntérképezésnek, valamint használatukkal egyedek könnyen és pontosan azonosíthatóak (Hidas, 2002).
Tipizálásuk annak a néhány bázispár ismétlődésének számbeli változékonyságán alapul, amelyek két, csak egy-egy adott genomrészletre jellemző szekvenciarészlet között helyezkednek el (Weber és May, 1989).
A mikroszatellitek ismert biológiai funkcióval nem rendelkező DNS szakaszok, amelyek szoros kapcsolatban állhatnak mennyiségi tulajdonságokat hordozó (létrehozó) poligénekkel (Komlósi és mtsai, 2005). Ezek a DNS szakaszok, mikroszatellitek jól felhasználhatók a mennyiségi tulajdonságokat meghatározó nagyhatású gének nyomon követésében, származásellenőrzésekben, a különböző populációszerkezeti vizsgálatokban (MacHugh és mtsai, 1994, 1997, 1998; Moazami-Goudarzi és mtsai, 1997;
Schmid és mtsai, 1999; Martin-Burriel és mtsai, 1999; Arranz és mtsai, 1998;
Saitbekova és mtsai, 1999; Canon és mtsai, 2000, Józsa és mtsai, 2006a). A DNS vizsgálatok különböző adataira támaszkodva olyan megoldások találhatók, amelyek lehetővé teszik a heterozigozitás, a genetikai variabilitás fenntartását kis létszámú állományokban. A decentralizált nukleusz tenyészetek közötti hasonlóságot vagy azonosságot is objektív módon tükrözik vissza (Mihók, 2002; Hansen és mtsai, 2002).
A mikroszatellitek viszonylag egyenletesen oszlanak el a genomban (Schlötterer és Pemberton, 1998), ami igen jó lehetőséget nyújt a
géntérképezéshez, a nagy marker variabilitást pedig igen jól lehet hasznosítani egyed ill. fajtaazonosításban és genetikai összehasonlításokban is (Takahashi és mtsai, 1998; Zhou és mtsai, 1999; Romanov és Weigend, 2001; Zsolnai és mtsai, 2006).
A molekuláris genetika lehetőségeinek hasznosítása integrált komponensévé kell váljék a hagyományos – szelekció és keresztezéses – nemesítési stratégiáknak. A hagyományosan alkalmazott cseppvérkeresztezés során egy fajta csak egy, vagy kevés számú monogénes tulajdonságban fejlesztendő. Markergének, mikroszatellitek felhasználásával kimutathatók azok az egyedek, amelyek a kívánatos gént nagy valószínűséggel hordozzák, így átadhatják a javítandó fajtának. Folyamatos visszakeresztezés és szelekció hatására, illetve után az ivadékokban becsülhető a reménykeltő gének jelenléte.
Ugyanezen az elven működik a fajtafenntartás során az alappopulációra jellemző génkészlet megmaradásának kimutatása (Mihók, 2002).
Az ismétlődések határoló szekvenciái specifikusak. Ezen határoló egyedi szekvenciákra tervezve primereket (szintetikusan előállított DNS szálak, a sokszorosítandó DNS szakasz végeinek komplementerei) lehetővé válik a mikroszatellitek ún. polimeráz láncreakcióval (PCR) történő vizsgálata (Fésüs és mtsai, 1997). A vizsgálatokhoz kis mennyiségű vérből, szövetből, szőrhagymából vagy nyálból izolált DNS minta szükséges (Bruford és Wayne, 1993).
A mikroszatellit vizsgálatok előnye például a RAPD (Random Amplified Polymorphic DNA = véletlen amplifikált polimorf DNS) módszerrel szemben, hogy egy lókuszon a heterozigócia is detektálható (kodominancia) és több allél is előfordulhat. Viszonylagos hátránya, hogy többé-kevésbé fajspecifikusak, ill.
csak részlegesen használhatók a közel rokon fajoknál is. Az egyes lókuszokra specifikus primerszekvenciákat kell tervezni, aminek előfeltétele természetesen, hogy szekvencia ismerettel rendelkezzünk a genom mikroszatelliteket hordozó fragmentjeiről. Ezért a mikroszatellit markerek kifejlesztése jelentős előmunkálatokat igényel. További hátrányai a módszernek, hogy nagy értékű szekvenáló elektroforézises készülékeken lehet csak hatékonyan vizsgálni őket, mivel a különböző allélek nem ritkán csak 1-4 bázispárban térnek el egymástól ill. az allélek pontos beazonosítása is ilyen felbontású futtatást igényel. Költségesebb ez a vizsgálat azért is, mert egy PCR
reakcióban csak egy lókuszra nyerünk információt. Lehetséges ugyan multiplex PCR alkalmazása is, több lókusz amplifikálása közös elegyben és protokollal, ez azonban újabb további optimalizálásokat igényel (Hidas, 2002).
3.1.3. PCR – polimeráz láncreakció (polymerase chain reaction) 3.1.3.1. A PCR sikertörténete
A tudománytörténet úgy tartja, hogy a polimeráz láncreakció (PCR:
Polymerase Chain Reaction) elve 1983 áprilisában Kary Banks Mullis elméjében született meg. A módszer elvét, egy gyakorlati eredmény bemutatásán keresztül, 1985-ben közölték munkatársaival a Science magazinban (Saiki és mtsai, 1985). Ettől az időponttól a PCR által, egy egyszerű elven alapuló, de zseniális technikával bővült a molekuláris biológia eszköztára. Ez a ciklikus, in vitro, enzimkatalizált, DNS-szintetizáló eljárás lehetőséget teremtett arra, hogy kimutatható szintre növeljék a vizsgálni kívánt DNS-szakaszt, lényegében megszüntetve így a DNS vizsgálatok mennyiségi korlátait. Jelenleg az élettudományok szinte minden ágában alkalmazzák ezt a technikát. A PCR-rel kapcsolatos termékek, reagensek és készülékek hatalmas tárháza áll ma már rendelkezésünkre, s ma már egy PCR készülék megszokott alapfelszereltsége egy molekuláris biológiai laboratóriumnak.
3.1.3.2. A polimeráz láncreakció elve
Ez a forradalmian új technika tehát egy olyan in vitro (egyes speciális alkalmazásokban in situ) módszer, amelynek során meghatározott DNS szakaszokat lehet enzimatikus módon megsokszorozni (amplifikálás) két, az amplifikálandó szakaszt közrefogó, specifikus szekvenciájú oligonukleotid (primer) segítségével (Mullis és Faloona, 1987). A PCR elve a DNS függő DNS polimerázok aktivitásán alapul. Ezek az enzimek egy indító oligonukleotid, a primer segítségével képesek lemásolni, és így megkettőzni az eredeti DNS szálat. Ismételt működésük során az eredeti DNS molekula, a templát hatványozottan felsokszorozódik (Innis és mtsai, 1990). Emiatt kapta ez a módszer a polimeráz láncreakció elnevezést. Akár 10000 bp (bázispárban határozzák meg a DNS hosszát) DNS-szakasz is felsokszorosítható ezzel a módszerrel. A PCR technika létrejöttét egy termostabil DNS-polimeráz, a Taq-
polimeráz felfedezése és az automatizált fűtőblokkok (PCR készülék, thermocycler) kialakítása tette lehetővé, mivel a reakció általában 50-95°C-os hőmérsékleti tartományban zajló ciklikusan ismétlődő lépésekből áll (Brock és Freeze, 1969). Korábban a PCR-hez az E. coliból származó Klenow enzimet használták. Ekkor azonban forraláskor nemcsak a DNS, hanem az enzim is denaturálódik, ami azt jelenti, hogy a hőérzékeny polimeráz enzimeket minden ciklus után újra kell mérni a reakcióba. Az amerikai Yellowstone Nemzeti park forró vizű hőforrásában fedezték fel a Thermus acquaticus baktériumot (Brock és Freeze, 1969), amely 94°C-on él és a belőle kinyert DNS polimeráz, a Taq DNS polimeráz ezen a hőmérsékleten is megőrzi aktivitását.
Az exponenciálisan felsokszorozott DNS agaróz vagy poliakrilamid- gélen mutatható ki, interkalálódó DNS festék (etidium-bromid) segítségével (Righetti, 1989; Lindstedt és mtsai, 2000).
3.1.3.3. A PCR reakció menete
Az amplifikáció során a DNS-szintézis, majd a képződött termék denaturációjának ismétlődő ciklusai (25–40 ciklus) zajlanak. A PCR reakció rendkívül érzékeny, elméletileg alkalmas arra, hogy akár egyetlen DNS molekulát is felszaporítson, és ez által vizsgálhatóvá tegyen (Mullis és Faloona, 1987; Saiki és mtsai, 1985). A sokszorosítani kívánt DNS-szakasz bázissorrendjét, szekvenciáját nem feltétlenül szükséges teljes egészében ismernünk (egyes speciális esetektől eltekintve), általában a két végén elhelyezkedő 15-30 nukleiotid sorrendjét kell tudnunk, ahová a primerek bekötődnek. A primerek jelölik ki az amplifikálni kívánt DNS-szakaszt. A kettősszálú DNS szétválása, denaturációja után történik a primerek hibridizációja (anneálás). A primerek a denaturált, egyszálú templát DNS lánchoz H-hidak által kapcsolódnak a bázispárosodás elve szerint, azaz kizárólag adenin-timin (A-T), citozin-guanin (C-G) kapcsolatok jönnek létre. A primer utolsó nukleotidjában szereplő dezoxiribóz 3’ helyén egy szabad OH csoport található, amelyhez a DNS függő DNS-polimeráz kapcsolja a soron következő komplementer nukleotod 5’ foszfát csoportját. A polimerizáció a primer 3’ végén kezdődik és addig tart, amíg véget nem ér a templát, vagy míg a DNS polimeráz le nem válik a megszintetizált kettősszálú DNS darabról (Dieffenbach és mtsai, 1995). Adott hosszúságú DNS szakaszok
amplifikációjához legalább két primert használunk a PCR reakcióban, az egyik meghatározza a kierősítendő szekvencia elejét (forward primer), amely az úgynevezett sense DNS-szálhoz hibridizál, a másik pedig a végét (reverse primer), amely az antisense DNS-szálhoz kötődik a reakció során. Így a reakció végén csak a kívánt DNS-régió lesz jelen látható mennyiségben.
Ha mindkét primer szelektíven tapad a megfelelő egyszálú templát komplementer részéhez és az elongációk során az újonnan szintetizált DNS szálak túlnyúlnak az „ellenprimer” tapadási helyén, akkor ezen szálak is célszekvenciaként (template) fognak szolgálni, növelve a rendelkezésre álló templát mennyiségét ciklusról ciklusra. A primerekkel kiválasztott DNS régió mennyiségi növekedése a következő formulával fejezhető ki: N=(2n-2n)x, ahol
„n” a ciklusok száma, „x” a kiindulási templát mennyiség kópiáinak száma,
„N” az n ciklus után jelenlevő amplifikált DNS kópiáinak a száma (Rolfs és mtsai, 1992).
A PCR reakció egy ciklusa az alábbi három lépésből áll:
A PCR lépései:
A reakció egy ciklusa 3 eltérő hőmérsékleten lejátszódó, 3 különböző lépésből tevődik össze (2. ábra; Zsolnai, 2000). A reakció statisztikai folyamatként fogható fel, ezért pl. az annealing hőmérsékleten is lejátszódik elongáció és viszont.
- denaturáció: magas hőmérsékleten (92-96C-on) a két DNS szál elválik egymástól, lehetővé téve, hogy a primerek majd kapcsolódhassanak rájuk. A folyamat ideje: 30 sec.
- annealing (primerek kötődése): 50-70C-on a primerek kötődnek az egyszálúvá alakított DNS komplementer szakaszához. Az annealási hőmérséklet elsősorban a primerek olvadási hőmérsékletétől függ. A folyamat ideje: 30 sec.
- elongáció (lánchosszabítás): ebben a szakaszban játszódik le az új lánc szintézise a már láncindító szekvenciaként viselkedő primer tökéletesen
feltapadó 3’ végéről (72-73C-on). A lépés időigénye egyrészt függ magától a DNS-polimeráztól, másrészt az amplifikálandó DNS-szakasz hosszától.
2. ábra: A PCR-ciklus sematikus ábrája.
(1) Denaturálás 92-96 °C-on. (2) Kapcsolódás 50-70 °C-on. (3) Meghosszabbítás 72-73 °C-on (P=polimeráz). (4) Az első ciklus véget ért. Az eredményül kapott két DNS-szál a következő ciklus templátja lesz, azaz minden ciklusban megkétszereződik a DNS mennyisége.
(forrás: http://hu.wikipedia.org/wiki/K%C3%A9p:Pcr.png)
3.1.3.4. A PCR reakcióelegy összetevői
- Templát vagy target DNS: az a DNS, amelyről nagyszámú másolatot akarunk készíteni; biológiai mintából izolált DNS, amely lehet genomiális- vagy
mitokondriális DNS vagy RNS-ről reverz transzkripcióval átírt úgynevezett cDNS
- primerek: szintetikusan előállított DNS szálak (oligonukleotidok); a primer 1 (forward) az egyik DNS szálon komplementer és az amplifikálódó DNS kiindulását definiálja; a primer 2 (reverse) a másik DNS szálon komplementer és a termék végét határozza meg
- dNTP-k: az új komplementer lánc felépítéséhez szükséges alkotóelemek (d:
deoxy, N: adenin – citozin - guanin - timin, TP: tri-foszfát)
- Hőstabil polimeráz (pl.:Taq): a nukleotidok beépítését (lánchosszabítást) végző enzim
- Puffer: a hőstabil polimeráz megfelelő működéséhez biztosítja az ideális pH- t, ionerősséget és sókoncentrációt
- MgCl2: a Taq polimeráz működéséhez nélkülözhetetlen; az optimális Mg2+
koncentráció általában 0,5-3 mM között mozog
- desztillált víz
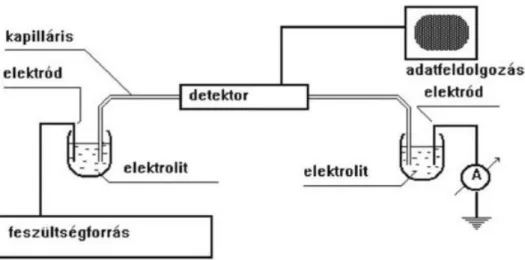
3.1.4. A kapilláris gélelektroforézis
A kapilláris elektroforézis egy olyan, napjainkban rendkívül gyorsan fejlődő analitikai elválasztási módszer, amely egyesíti a klasszikus elektroforézis technikáját a modern kromatográfiás detektálás és automatizálás műszeres lehetőségeivel (3. ábra; Issaq, 2000). Az elektroforetikus elválasztási módszerek azon alapulnak, hogy elektromos térben az oldott anyagok különböző sebességgel vándorolnak méretük és/vagy töltésük függvényékben (Tiselius, 1937). A kapilláris elektroforézisnél (capillary electrophoresis, CE) az elválasztás egy vékony, általában 25-75 μm belső átmérőjű, puffer oldattal töltött 35-65 cm hosszú kapillárisban történik. Az elektroforézist kis átmérőjű kapillárisban kivitelezve lehetővé válik nagy elektromos mező alkalmazása,
mert a kis kapilláris hatásosan adja le a keletkező hőt. Az elektromos mező növelésével hatékonyabb elválasztás érhető el rövidebb idő alatt. Az alkalmazott feszültség 10-30 kV. A kapilláris elektroforézisnél a csatorna falát úgy képezik ki, hogy az valamilyen (általában negatív) elektromos töltésű csoportokat tartalmazzon, vagy a megfelelő pH-n negatív töltésűvé váljék, (de nemionos anyagok is kialakíthatnak EEO-t a felületükre adszorbeálódott anionoknak köszönhetően). Ebben az esetben az oldatban lévő kationok a negatív töltésű falhoz vándorolnak, ezzel a csatorna felületén elektromos kettősréteget alakítanak ki. A csatorna két végére elektromos feszültséget kapcsolva a felületen lévő szolvatált kationok elmozdulnak a negatív töltésű elektród irányába, és ezzel mozgásba hozzák a csatornában levő oldatot. Mivel az EEO jelentősen nagyobb az anionok effektív mobilitásánál, ezért nemcsak a kationok vándorolnak, hanem az anionok is. Így az anionok és kationok egyaránt egy futtatás alatt analizálhatók. A töltés nélküli részecskék nem választhatóak el egymástól. Az elválasztás során fontos, hogy az EEO-t megfelelően szabályozzuk (Engelhardt és mtsai, 1996).
Az elektroforézist, mint elválasztási technikát Tiselius 1937-ben vezette be (Tiselius, 1937) amikor különböző fehérjéket tartalmazó oldatot helyezett egy, a végeinél puffer oldatokkal érintkező csőbe, és elektromos feszültséget kapcsolt a cső végeire, azt találta, hogy a minta komponensei töltésüktől és mozgékonyságuktól függően különböző sebességgel különböző irányokba vándoroltak. Munkájáért Tiseliusnak később Nobel-díjat adományoztak.
A kapilláris gélelektroforézis (capillary gel electrophoresis CGE) a hagyományos lap-gél elektroforézis kapilláris elektroforézis megfelelője.
Biológiai makromolekulák (oligonukleotidok, DNS fragmensek és fehérjék) méreten alapuló elválasztására használják (Smith, 1991; Swerdlow és Gesteland, 1990). Főbb előnyei a lap-gél elektroforézissel szemben, hogy sokféle gél-mátrix használható és széles tartományban változhat az összetétel.
A kapillárist géllel töltve az megakadályozza az elektroozmotikus áramlást.
Ekkor az elválasztás ugyanúgy történik, mint a hagyományos gélelektroforézisnél, de a kapilláris miatt jobb felbontás és nagyobb érzékenység érhető el, valamint lehetővé válik az on-line detektálás. Az elektroforézis szelektivitása nagy molekulatömegű anyagok elválasztásakor nő,
ha a kapillárist a kapilláris hosszában folytonosan változó pórusármérőjű géllel töltjük meg. Az elektromos erőtér hatására a töltéssel rendelkező molekulák a töltéssűrűség (töltés/ionsugár) alapján eltérő sebességgel vándorolnak, amelyet az ún. molekula szűrőhatás megváltoztat. Érdemes megemlíteni, hogy a töltéssűrűség, a forma és tömeg különbségek elválasztásra történő kihasználásánál a DNS szeparálás esetében szinte kizárólag a tömeg különbség kerül kihasználásra. A kapillárisba poliakrilamidot, agarózt vagy egyéb géleket tölthetünk. Elsősorban fehérjék és nukleinsavak elválasztására használjuk a poliakrilamid (PAGE) és Na-dodecil-szulfát (SDS) töltetet (SDS-PAGE). A kvarckapillárisban a gél polimerizációját kémiai módszerrel vagy gamma- sugárzással végzik. A gél lehet térhálós (3 dimenziós szerkezetű) vagy lineáris.
Sok esetben megfelelő szelektivitás érhető el, ha a kapillárist polimer oldattal töltjük. A töltött csőben az elektroozmózis okozta anyagvándorlás megszűnik.
Gélelektroforézissel csak töltéssel rendelkező molekulák elválasztását tudjuk megoldani (Swerdlow és Gesteland, 1990).
3. ábra: A kapilláris elektroforetikus készülék sematikus rajza
(forrás: http://www.hik.hu/tankonyvtar/site/books/b154/ch09s04s02.html)
A kapilláris gélelektroforézis jól automatizálható, a gép automatikusan felveszi a DNS mintát, majd méret alapján elvégzi a szétválasztást.
Fluoreszcens jelölés esetén az egyes mikroszatellitek azonosíthatók, és a műszer elvégzi a méret-meghatározást is.
3.2. Molekuláris genetika az állattenyésztésben
A XX. század elején gazdasági állatok esetében intenzív kutatómunka kezdődött a vércsoportok, a biokémiai polimorf rendszerek területén. Az ötvenes évektől e vizsgálatok eredményeit felhasználták az állattenyésztési gyakorlatban, elsősorban a szülői származásellenőrzésben (Csontos és mtsai, 1993). A származásellenőrzéseknél az ún. kizárásos módszert alkalmazzák, melyet a humán igazságügyi gyakorlatból vettek át. A módszer lényege:
meghatározott immuno-genetikai tulajdonságokra megvizsgálják a szülők (apa, anya), valamint az utód vérét. Az utód egyik szülőjét (ált. anya) biztosnak tekintik. Amennyiben az utód vérében olyan faktorokat találnak, ami a biztosnak mondott (hitt) szülő vérében nincs meg, azt csak a másik szülőjétől örökölhette. Ha az utód ezen tulajdonságai a másik szülőben nem mutathatóak ki, az utód származása attól a szülőtől kizárt (Flink és Csontos, 1988). A vizsgálatok megbízhatósága nagymértékben függ a vizsgált faktorok számától.
A származásellenőrzés vizsgálatok mellett folyamatosan publikálták az első olyan kutatások eredményeit, amelyekben megkíséreltek összefüggést találni értékmérő tulajdonságok (termelési, szaporodási tulajdonságok) és az öröklődő vércsoport és a biokémiai polimorfizmus tulajdonságok, mint markerek között (Soós, 1986). Ezen a területen igazi előretörés napjainkban tapasztalható, amikor a molekuláris genetikai ismeretek alkalmazása révén olyan DNS szintű polimorfizmusokat ismerhetünk meg, amelyek számos területen felhasználhatók az állattenyésztési gyakorlatban (Fésüs és mtsai, 2000).
Hazánkban a vércsoport és a vér biokémiai polimorfizmusok vizsgálatára kidolgozott módszereket alkalmazták a populációk közötti, illetve a populáción belüli távolság, diverzitás meghatározására az állattenyésztésben (Dohy, 1979;
1999; Cothran és Kovac, 1997; Nozawa és mtsai, 1998; Józsa és mtsai, 2006b).
A fajtarokonsági viszonyok illetve a fajták közötti genetikai távolságok meghatározhatóak, ha összehasonlítjuk az egyes fajtákban kapott vércsoport-, szérumfehérje- és enzimgének gyakorisági értékeit. A kapott összefüggések a fajták fejlődéstörténetében való evolució eredményeit azonosíthatják. A fajták közötti genetikai távolságok ismerete különösen hasznos a fajtakeresztezések megtervezésekor és elősegítik a heterózishatásnak becslését (Fésüs, 1984).
Megbízhatóbb eredményeket, szélesebb körben való alkalmazhatóságot
azonban csak a DNS szintű vizsgálatok jelentik. A populációgenetikai vizsgálatok során a DNS mikroszatellit vizsgálatok segítségével kifejezhető a genetikai variabilitás számszerűen is, becsülhető egy adott állomány beltenyésztettségének (homozigozitásának) mértéke is. Ennek elméleti alapja az, hogy a beltenyésztettség mértéke arányos a homozigóta egyedek adott állományon belüli viszonylagos gyakoriságával (Fésüs, 1984).
Az utóbbi évtizedben a molekuláris genetika területén végbemenő fejlődés számos DNS marker felfedezését és kimutatásukra alkalmas módszerek kidolgozását jelentette (Achmann és mtsai, 2000; Woodruff, 1993).
3.2.1. Genetikai markerek alkalmazása az állattenyésztésben
A kódolt és nem kódolt genomi szakaszok variabilitásának a megismerésével létrejött a genetikai markerek fogalma (Jeffreys és mtsai, 1985; Weber és May, 1989). Az ideális genetikai marker nagymértékben polimorf, allélváltozatait egyértelműen, gyorsan és egyszerűen lehet kimutatni, továbbá előnyös, ha belőlük minél többet sikerül kimutatni a genom bármely területén (Dohy, 1999). A legrégebbi genetikai markerek látható genetikai jegyek voltak (pl: szőrszín, szarv megléte, hiánya), továbbá enzimpolimorfizmusok (pl: transzferrin, albumin), vércsoportok (pl: ló esetében D vércsoport, szarvasmarha esetében B vércsoport) stb. Ezek a hagyományos markerek sem polimorfizmusuk tekintetében, sem számukban nem voltak elégségesek átfogó genetikai térképezés végrehajtására. A rekombináns DNS technika megjelenésével fedezték fel a molekuláris genetikai markereket, amelyek döntő hányada génektől független, polimorfizmust mutató DNS szakaszok (Hardge, 1999). A biokémiai és immunológiai markerekkel szemben számos előnyük van. Elviekben minden szövetből, az egyed korától, nemétől függetlenül mód van DNS izolálásra.
Genomon belüli eloszlásuknak, illetve a magasfokú polimorfizmusuknak köszönhetően (egy marker akkor számít polimorfnak, ha egy populáción belül minimum két allélja van, aminek a gyakorisága nincs 1 % alatt (Harge, 1999) egyre jobban előtérbe kerültek a populációgenetikai tanulmányokban (Hedrick és Miller, 1992, Russel és mtsai, 1993; Van Zeveren és mtsai, 1995; Magoulas, 1998). A molekuláris genetikai markereket O’brien (1991) két csoportra, I. és
II. típusú markerekre osztotta. Az I. típusú markerek maguk a gének, amelyek kódolt funkcióval rendelkeznek. A fehérjéket kódoló területeknek azonban alacsony a mutációs rátájuk, mert az életfontosságú fehérjékműködésének a megőrzéséhez ez elengedhetetlen feltétel. A II. típusú markereknek nagyobb variabilitása van (Litt és Luty, 1989; Weber és May, 1989; Tautz, 1989). Ebbe a csoportba tartoznak a mikroszatellit markerek is.
Az állattenyésztésben és a növénytermesztésben a termelési értékmérők alakulásában fontos szerepet játszó gének, génváltozatok markereinek keresése a fő cél, hogy ezek azonosításával megvalósulhasson a markereken alapuló szelekció. Ez a kutatómunka rengeteg DNS markert tárt fel különböző fajokban, amelyek kisebb-nagyobb mérvű polimorfizmust mutatnak és igen alkalmasak egyedek, állományok genotipizálására. Különösen jelentősek a nem aktív génekben előforduló polimorfizmusok, mivel ezek nincsenek szelekciós hatás alatt, mutációval történő változásai neutrálisak a fenotípusra ill. a túlélésre, ezért fennmaradnak a generációk során (Hidas, 2002).
A molekuláris markerekkel számos kalkulációt végezhetünk és gyakoriságuk puszta megállapítása mellett további információkat nyerhetünk az állományokon belüli értékelésekben, pl: az állomány homogenitása, heterozigozitás, genetikai diverzitás, genetikai változatosság (Zhu és mtsai, 1996a; Machado és mtsai, 2003; Maudet és mtsai, 2002; Bjornstad és mtsai, 2000; Tozaki és mtsai, 2003, Achmann és mtsai, 2004; Cothran és mtsai, 1987;
Korom és mtsai, 2003). Az állományok összehasonlítására a genetikai távolság (hasonlóság) becslése az alkalmas megközelítés (Nei, 1972). Ezek mellett nagy jelentősége van a molekuláris martkereknek az állományok, populációk beltenyésztettségi szintjének megállapításában is (Curik és mtsai, 2003; Zhu és mtsai, 1996b; Cunningham és mtsai, 2001; MacHugh és mtsai, 1998).
Különösen a génmegőrzésben szerepet játszó állományoknál nagy jelentősége lenne az erre vonatkozó információknak.
3.2.2. Molekuláris genetika a génmegőrzésben
A molekuláris genetikai analízisek révén nyert információk génmegőrzésben való közvetlen használata körül ma még nincs teljes konszenzus. Kezdetben a genetikai távolságok számításával értékelték a
fajtákat, ami azt jelentette, hogy génmegőrzési szempontból azok a legértékesebb fajták, amelyek a többitől legjobban eltérnek a markerek tekintetében. Újabb keletű szempont a megőrzendő állományok belső genetikai szerkezete is, azaz mekkora méretű variabilitást mutatnak a populációk. Ez a mutató természetesen jelentősen függ a fajta állományának történetétől, előfordult-e jelentős létszámcsökkenés a teljes állományt vagy csak a használt apaállatok számát illetően. Az aktuálisan mutatott genetikai variabilitás ezért jobban függ a fajta tenyésztésének múltjától, mint a jelenlegi állománymérettől. A fajták további értékelési szempontjai lehetnek a fajták ismert értékmérői, különleges, egyedi tulajdonságai (rezisztencia, húsminőség, stb.), amelyek természetesen magasabb értéket kölcsönöznek a fajtának (Hidas, 2002).
A génmegőrzés célja, hogy a populációkat lehetőleg változatlan formában tartsuk fent. Ismeretes, hogy szelekció nélkül nem lehet tenyészteni, hiszen nem hagyunk meg minden állatot továbbtenyésztésre. Rendkívül fontos, hogy a ritka, kislétszámban előforduló értékeket megőrizzük. Ennek érdekében igyekszenek megőrizni azokat a genetikai markereket (vércsoport, polimorf allélek, mikroszatellit), amelyeknek csak néhány hordozója van. Ez akkor is érvényes, ha nem tudjuk, hogy milyen tulajdonságokkal vannak összefüggésben ezek a markerek (Takács és mtsai, 2006). Magyarországon több kutató is célul tűzte ki e nemes feladatot. Hazánkban számos munka jelent meg őshonos magyar fajták molekuláris genetikai felmérésére DNS mikroszatellit markerek segítségével (Takács és mtsai, 2006; Hidas és mtsai, 2006; Mihók és mtsai, 2006; Radnóczy és mtsai, 2006; Bán és mtsai, 2006;
Kusza és mtsai, 2006).
A géntartalékok védelmének célja a kihalástól veszélyeztetett háziállatfajták megmentése. A fajta viszont több mint csupán annak neve. A fajták védelmének értelme a jövő generációk ismeretlen igényeinek szolgálata, ezért a tulajdonságokat és az ezek alapját képező géneket kell megmenteni, nem csak a fajta nevét (Bodó, 2002). Sokszor azonban „nem könnyű igazságot tenni a hosszú időre szóló idealista tervek és a tenyésztők azonnali álmai között” (Alderson 1989).
Ezt a célt csak úgy lehet megvalósítani, ha a fajták tulajdonságait értékeljük és kutatjuk termékeik felhasználásának lehetőségét. „Leginkább az
teszi jogosulttá a fajták védelmét, hogy tulajdonságaiknak értéke a jövőben nőni fog. Következetes programra van szükség, hogy ezeket a tulajdonságokat megállapítsuk és hasznosítsuk” (Alderson, 1989).
3.2.3. Molekuláris genetika a származásellenőrzésben
Egy állat származásának ismerete az állattenyésztésben alapvető fontosságú. A tenyészérték, ezáltal a forgalmi érték meghatározása nagyon lényeges. Abban az esetben, ha a származás nem ismeretes, nem állapítható meg az utódok öröklött tulajdonságaiban a szülők befolyása, s így a fajta genetikai előrehaladásának meghatározása is akadályba ütközik (Geldmann és mtsai, 1986; Israel és Weller, 2000).
A származásellenőrzés területén a vércsoport és a vér biokémiai polimorfizmusainak vizsgálatára kidolgozott módszereket alkalmazták az állattenyésztésben (Bowling, 1996). A kizárásos módszer ténye, a vitás esetek tisztázatlansága, a nehézkes és hosszadalmas eljárás, valamint a minták szállításának, tárolásának problémái felvetettek egy megbízhatóbb és nagyobb hatékonyságú módszer kidolgozásának ill. adaptálásának szükségességét.
Mindezek mellett a kérdés azért is aktuális volt számunkra, mert egyre erőteljesebb törekvések irányulnak az Európai Unió tagállamaiban egy egységes származásellenőrzési módszer bevezetésére. Ez a világban folyó kutatások alapján a molekuláris genetikai módszerek közül került ki (Fésüs és mtsai, 1997).
A DNS vizsgálattal végzett származásellenőrzési módszerek magyarországi bevezetését indokolták és a következő hazai és nemzetközi történések tették szükségessé:
Az európai és a magyar állategészségügyi szabályok megváltozása - immunsavók termelésének leállítása
ISAG (Inernational Society for Animal Genetics) kongresszus 1998- ban meghatározta a vizsgálandó mikroszatelliteket ló és szarvasmarha fajban egyaránt
2000, az utolsó vércsoport és biokémiai polimorfizmus alapján végzett körtesztek éve
Megszűnik a vércsoport és biokémiai polimorfizmusok vizsgálatához használatos vizsgáló savók standardizálásának lehetősége
2000-től kizárólagos DNS alapú körteszt
A szarvasmarha faj esetén még néhány egyéb ok is indokolttá tette a DNS alapú származásellenőrzés bevezetését. Hazánkban a húsmarhák tartása legelőre alapozott, gulya-tartásban történik. Az ágazatban csekély mértékű a mesterséges termékenyítés alkalmazása, így a legelőn tartott állatok szabad- vagy háremszerű pároztatása a bevált módszer. Az utódok származásellenőrzése a borjak választása után történik: az anya általában ismert, de az apák esetében csak rendszerint azt tudjuk, hogy egy-egy időszakban mely bikák fedeztek a gulyában. Abban az esetben, ha a fajta genetikailag homogén, a fedezésre használt bikák genotípusa hasonló vagy azonos, a hagyományos vércsoport és biokémiai polimorfizmus vizsgálatok alapján a borjak nagy hányadának nem állapítható meg biztonsággal az apai származása. (A húsmarha egyedek kimutatható vércsoport faktorainak, és alléljeinek száma a tejelő-, és kettős hasznú fajtáénak kb 20-25 %-át teszi ki.) Ez vonatkozik főleg a hereford és angus fajtákra, mely áttételesen rontja a tenyészérték becslés megbízhatóságát is, ha az utódok apákra egyértelműen nem különíthetőek el.
3.2.3.1. A DNS–re alapozott származásellenőrzés módszerének főbb vonatkozásai
A DNS alapú származásellenőrzés vizsgálatok lényege, hogy egy diploid szervezet két homológ kromoszóma garnitúrával rendelkezik, ezért egy lókuszon két mikroszatellit allélt hordoz. Az allél ebben az esetben a nukleotid szekvenciák ismétlődésének a számával lesz kapcsolatban. Egy utód a kérdéses (mikroszatellit) lókuszon olyan két alléllel (szekvencia ismétlődési számmal) rendelkezhet, melyek közül az egyiket az anyától, a másikat az apától örökölte.
Ezért az utód minden egyes vizsgált mikroszatellit lókuszát össze kell hasonlítani a szülők megfelelő lókuszával (az adott mikroszatellit ismétlődési számával). Amennyiben minden vizsgált mikroszatellit esetében érvényes, hogy az utód mikroszatellit hossza az adott lókuszon a szülőként megjelölt
anyában és apában fellelhető, akkor ezzel a vizsgálattal a szülői származás
„nem zárható ki” (Mihók, 2002).
A mikroszatellit analízis során kapott eredményekből meghatározhatjuk az utód származását, azok ismerete hasznosan felhasználható a tenyésztésben.
Azokra épülő számos kalkulációval pedig populációgenetikai összefüggésekre következtethetünk (Juras és Cothran, 2004; Luís és mtsai, 2002; Sun-joung Lee és Gil-jae Cho, 2006; Tozaki és mtsai, 2001; Van Haeringen és Van Haeringen, 1992).
3.2.3.2. Származásellenőrzés Magyarországon
Magyarországon jelenleg nem mindegyik fajtatenyésztő egyesület (ló és szarvasmarha egyaránt) kötelezi tagjait a DNS alapú származásellenőrzés elvégzésére. Minden egyesület maga hozhat határozatot, hogy kéri-e az említett vizsgálatot, vagy sem. Ez alól a nemzetközi fajták jelentenek kivételt (pl. angol telivér).
3.2.3.2.1. Származásellenőrzés a lótenyésztésben
A ló faj esetében az angol telivér, az arab telivér, a lipicai, a magyar sportló, az ügető és a quarter horse fajtákban kötelező a DNS alapú származásellenőrzés, csak a vizsgálat elvégzésével törzskönyvezik a csikókat.
Ezenkívül a mesterséges termékenyítésből született csikókat kötelezően vizsgálni kell fajtától függetlenül. A kisbéri, gidrán, nóniusz, a póni, és kisló fajták esetében az adott évjárat véletlenszerűen kiválasztott 10 százaléka kerül vizsgálat alá. A furioso-north star és hidegvérű fajtákban nem kötelező a DNS alapú vizsgálat. Fajtától függetlenül azonban csak az az egyed válhat apaállattá, amelyik átesik az említett vizsgálaton, azaz minden ménjelölt DNS alapú származásellenőrzése kötelező.
Magyarországon a Mezőgazdasági Szakigazgatási Hivatal, Állattenyésztési Igazgatóság, Központi Állattenyésztési Laboratóriuma (volt OMMI) 2005. szeptember 15.-től csak DNS alapú származásellenőrzést végez a lovak esetében, néhány kivételtől eltekintve (pl.: anya-, ill. apaállat elpusztult, külföldön tartózkodik az állat stb.) A származással, tenyésztéssel kapcsolatos információkat az OLIR (Országos Lótenyésztési Információs Rendszer) adatbázisában nyomon lehet követni.
A vizsgálat elvégzéséhez szükséges vérmintát (speciális esetekben szőr illetve sperma) jelenleg az állatorvos a lótenyésztési felügyelő jelenlétében veheti le a lovaktól.
3.2.3.2.1. Származásellenőrzés a szarvasmarha tenyésztésben
A szarvasmarha faj esetén ugyancsak a tenyésztő egyesületek szabhatják meg tagjaiknak, hogy kérik-e a DNS alapú származásellenőrzés vizsgálatot, vagy sem. A tenyészállatok, illetve a tenyészbikák DNS vizsgálata azonban kötelező a 2. generációig (apa, anya is). Különleges esetekben (pl.: az egyed elpusztult, vagy nincs az országban) a Mezőgazdasági Szakigazgatási Hivatal, Állattenyésztési Igazgatóság, Központi Állattenyésztési Laboratóriuma (volt OMMI = Országos Mezőgazdasági Minősítő Intézet) vércsoport és biokémiai polimorf rendszerek vizsgálatát végzi el, DNS vizsgálat helyett.
A magyar szürkemarha fajta esetében vércsoport vizsgálatot is végez a laboratórium, a génfenntartás, a biodiverzitás és a különleges allélok meghatározása céljából. A bikák esetében itt is kötelező a DNS vizsgálat.
A húsmarha fajták (hereford, limousin, aberdeen angus) esetében számos egyesület a DNS alapú származásellenőrzést kérte, mivel jóval nagyobb pontossággal határozható meg a borjak származása, mint a vércsoport vizsgálatokkal (nagyon kevés a vércsoport allél).
2004. július 1.-től központi lajstromszámot csak az a bika kaphat, amelynek van DNS alapú vizsgálata. A törzskönyvezést több országos adatbázisban lehet nyomon követni (ENAR = Egységes Nyilvántartási és Azonosítási Rendszer, TER = Termékenyítés Ellenőrzési Rendszer adatbázisok).
A vizsgálat elvégzéséhez szükséges vérmintát, illetve szőrmintát jelenleg az állatorvos veszi az állatoktól, de a tulajdonos, vagy a tenyésztési vezető is küldhet be a laboratóriumba mintákat.
4. ANYAG ÉS MÓDSZER
4.1. A kísérleti minták gyűjtése és előkészítése a molekuláris genetikai vizsgálatokhoz
A vizsgálatokat az Mezőgazdasági Szakigazgatási Hivatal, Állattenyésztési Igazgatóság, Központi Állattenyésztési Laboratóriumában (Budapest) végeztem. A felhasznált minták a rutinlaborba származásellenőrzés céljából (DNS vizsgálat) kötelezően 2004-2006 között beküldött vérminták voltak, amelyeket a randomizálás elvén választottam ki mindkét faj (ló, szarvasmarha) esetében. A vizsgált egyedek vegyesen nő, illetve hím ivarúak voltak (apa, anya, utód).
A DNS-mikroszatellit vizsgálatokhoz vett vérmintákat a humán gyakorlatban is ismert EDTA tartalmú vérvételi csövekbe sterilen gyűjtöttem, a vizsgálatok elvégzéséig -20˚C-on tároltam. A minták azonosságát a munkafolyamatok során szigorúan és pontosan biztosítanom kellett. A felhasznált vegyszerek analitikai tisztaságúak és DNS-áz mentesek voltak.
Az ISAG (Inernational Society for Animal Genetics; ISAG Iroda:
Laboratoire de Génétique Biochimique et Cytogénétique, INRA-CRJ, 78350 JOUY-EN-JOSAS, Franciaország; weblap: http://www.isag.org.uk/) Társaság által, a lovak származásellenőrzésében kötelező 9 mikroszatellit mellett még 8 mikroszatellitet (Dimsoski, 2003) használtam vizsgálataimban, a szarvasmarha esetében pedig 11 mikroszatellitet (Glowatzki-Mullis és mtsai, 1995), amelyeket az 1. és a 2. táblázatok foglalnak össze.
1. táblázat: A ló fajban vizsgált 17 mikroszatellit listája (StockMarks® for Horses Equine 17-plex Genotyping System, Applied Biosystems)
Mikroszatellit Primer szekvencia (5’→ 3’) Mérettartomány
(bázispár) Referencia
VHL20 HTG4 AHT4 HMS7 AHT5 HMS6 HTG6 ASB23 ASB2 HTG10
HTG7 HMS3 HMS2 ASB17 LEX3 HMS1 CA425
(FAM)-CAAGTCCTCTTACTTGAAGACTAG AACTCAGGGAGAATCTTCCTCAG (FAM)-CTATCTCAGTCTTGATTGCAGGAC
CTCCCTCCCTCCCTCTGTTCTC (FAM)-AACCGCCTGAGCAAGGAAGT
GCTCCCAGAGAGTTTACCCT (FAM)-CAGGAAACTCATGTTGATACCATC TGTTGTTGAAACATACCTTGACTGT
(VIC)-ACGGACACATCCCTGCCTGC GCAGGCTAAGGGGGCTCAGC (VIC)-GAAGCTGCCAGTATTCAACCATTG CTCCATCTTGTGAAGTGTAACTCA (VIC)-CCTGCTTGGAGGCTGTGATAAGAT GTTCACTGAATGTCAAATTCTGCT
(VIC)-GCAAGGATGAAGAGGGCAGC CTGGTGGGTTAGATGAGAAGTC (VIC)-CCACTAAGTGTCGTTTCAGAAGG CACAACTGAGTTCTCTGATAGG (NED)-CAATTCCCGCCCCACCCCCGGCA
TTTTTATTCTGATCTGTCACATTT (NED)-CCTGAAGCAGAACATCCCTCCTTG ATAAAGTGTCTGGGCAGAGCTGCT (NED)-CCAACTCTTTGTCACATAACAAGA CCATCCTCACTTTTTCACTTTGTT (NED)-CTTGCAGTCGAATGTGTATTAAAT
ACGGTGGCAACTGCCAAGGAAG (PET)-GAGGGCGGTACCTTTGTACC ACCAGTCAGGATCTCCACCG (PET)-ACACTCTAACCAGTGCTGAGACT GAAGGAAAAAAAGGAGGAAGAC
(PET)-CATCACTCTTCATGTCTGCTTGG TTGACATAAATGCTTATCCTATGGC
(PET)-AGCTGCCTCGTTAATTCA CTCATGTCCGCTTGTCTC
86-105 120-140 146-170 170-188 129-149 157-171 80-107 176-212 240-270 83-110 114-128 149-172 215-236 89-131 137-160 166-178 224-247
van Haeringen és mtsai. (1994) Ellegren és mtsai. (1992)
Binns és mtsai. (1995) Guérin és mtsai. (1994) Binns és mtsai. (1995) Guérin és mtsai. (1994) Ellegren és mtsai. (1992)
Irvin és mtsai. (1998) Breen és mtsai. (1997) Marklund és mtsai. (1994) Marklund és mtsai. (1994) Guérin és mtsai. (1994) Guérin és mtsai. (1994) Breen és mtsai. (1997) Coogle és mtsai. (1996) Guérin és mtsai. (1994) Eggleston-Scott és mtsai. (1997)