PANNON EGYETEM GEORGIKON KAR
Állattudományi és Állattenyésztéstani Tanszék
DOKTORI (PhD) ÉRTEKEZÉS
Készült a Pannon Egyetem, Állat- és Agrárkörnyezet- tudományi Doktori Iskola keretében
Doktori Iskola vezető:
Dr. ANDA ANGÉLA, DSc egyetemi tanár
TEJTERMELÉST ÉS HÚSMINŐSÉGET BEFOLYÁSOLÓ DGAT1 K232A, LEPTIN C528T, TG 5’ UTR
POLIMORFIZMUSOK VIZSGÁLATA HAZAI SZARVASMARHA POPULÁCIÓKBAN
Témavezető:
Dr. SZABÓ FERENC, DSc egyetemi tanár
Társ-témavezető:
Dr. ANTON ISTVÁN, PhD tudományos főmunkatárs
Készítette:
FARKAS VALÉRIA
KESZTHELY
2012
TEJTERMELÉST ÉS HÚSMINŐSÉGET BEFOLYÁSOLÓ DGAT1 K232A, LEPTIN C528T, TG 5’ UTR POLIMORFIZMUSOK VIZSGÁLATA HAZAI
SZARVASMARHA POPULÁCIÓKBAN
Értekezés doktori (PhD) fokozat elnyerése érdekében Írta:
FARKAS VALÉRIA
Készült a Pannon Egyetem Állat- és Agrárkörnyezet-tudományi Doktori Iskola iskolája keretében
Témavezető: DR. SZABÓ FERENC, DSc, egyetemi tanár, az MTA doktora Elfogadásra javaslom (igen / nem)
……….
(aláírás)
Társ-témavezető: DR. ANTON ISTVÁN, PhD Elfogadásra javaslom (igen / nem)
……….
(aláírás) A jelölt a doktori szigorlaton ...%-ot ért el,
Az értekezést bírálóként elfogadásra javaslom:
Bíráló neve: …... …... igen /nem
……….
(aláírás) Bíráló neve: …... …...igen /nem
……….
(aláírás) Bíráló neve: …... …... igen /nem
……….
(aláírás) A jelölt az értekezés nyilvános vitáján …...%-ot ért el.
Keszthely, 2011………. ……….
a Bíráló Bizottság elnöke A doktori (PhD) oklevél minősítése…...
………
Az EDHT elnöke
KIVONAT
A szerző 4 hazai szarvasmarha populációhoz tartozó (magyar tarka: 485; holstein- fríz: 417; jersey: 341; angus: 80), 7 állomány (összesen: 1323 egyed) DGAT1 (diacilglicerol -O- aciltranszferáz) K232A, leptin C528T és TG (tireoglobulin) 5’ UTR polimorfizmus vizsgálatát végezte el. A vér-, illetve szőrminták tipizálásához, a DGAT1 K232A és leptin C528T polimorfizmusok esetében Real-time PCR, TaqMan allélikus diszkriminációs módszert, míg a TG 5’ UTR polimorfizmus esetében PCR-RFLP módszert alkalmazott. A real-time PCR, valamint a PCR-RFLP módszerek segítségével mindhárom lókusz vizsgálata során 3 különböző mintázatot (genotípust) különített el (DGAT1:
AA/AA, AA/GC, GC/GC; leptin: CC, CT, TT; TG: CC, CT, TT).
A genotipizálást követően allélgyakorisági és populációgenetikai számításokat, valamint a vizsgált lókuszok (DGAT1 K232A, leptin C528T és TG 5’ UTR) és a termelési adatok közötti összefüggésvizsgálatot végezte el (SPSS 14.0 for Windows).
Vizsgálatai során szignifikáns kapcsolatot mutatott ki a három hazai tejelő szarvasmarha populáció (magyar tarka, holstein-fríz, jersey) DGAT1 K232A, leptin C528T és TG 5’
UTR genotípusok, valamint a tejtermelési paraméterek között.
A vizsgált húshasznú angus szarvasmarha populáció esetében szignifikáns kapcsolatot mutatott ki a DGAT1 K232A, leptin C528T és TG 5’ UTR genotípusok, valamint a hosszú hátizom (musculus longissimus dorsi: LD) és fehérpecsenye (musculus semitendinosus:
ST) intramuszkuláris zsírtartalma között.
ABSTRACT
STUDY OF THE EFFECT OF DGAT1 K232A, LEPTIN C528T, TG 5’ UTR POLYMORPHISMS ON MILK PRODUCTION AND MEAT QUALITY IN
HUNGARIAN CATTLE POPULATIONS
The autor estimate the effect of different Hungarian breeds of cows (Hungarian Simmental: 485, Holstein Friesian: 417, Jersey: 341 and Angus: 80) from seven herd (all:
1323), and DGAT1 K232A (diacylglycerol -O- acyltransferase 1) leptin C528T and TG 5’
UTR (thyroglobulin) loci. The blood and hair samples were typed for the bovine DGAT1 K232A and leptin C528T gene using a TaqMan allelic discrimination method in a Rotor- Gene RG 3000 Real-Time PCR system, for the TG 5’ UTR gene using PCR-RFLP method. As well Real-Time PCR system as PCR-RFLP method were all tree locus tree different alleles determined (DGAT1: AA/AA, AA/GC, GC/GC; leptin: CC, CT, TT; TG:
CC, CT, TT).
After genotyping the allele frequencies and population genetics calculations were carried multivariantanalyses (by SPSS 14.0 software) out to evaluate the relationship between genotype and milk and meat production.
Statistical results show signifant relationship between three Hungarian dairy cattle breeds population (Hungarian Simmental, Holstein Friesian, Jersey) DGAT1 K232A, leptin C528T and TG 5’ UTR genotypes and milk production.
The examine for Angus bulls population show signifant relationship between DGAT1 K232A, leptin C528T and TG 5’ UTR genotypes and fat percentage values in the musculus longissimus dorsi (LD) and musculus semitendinosus (ST).
AUSZUG
UNTERSUCHUNG VON DGAT1 K232A, LEPTIN C528T, TG 5’ UTR POLYMORPHYSMUS MIT AUSWIRKUNG AUF MILCHPRODUKTION UND
FLESICHQUALITAT IN UNGARISCHEN RINDERPOPULATIONEN
Die Autorin testete in seiner Arbeit vier verschiedene ungarische Rinderpopulationen, insgesamt 1323 Kühe (Ungarisches Fleckvieh 485, Holstein-Friesian 417, Jersey 341 und Angus 80) in sieben Betrieben. Die geprüfte Polymorphysmus-Typen waren: DGAT1 K232A (diacylglycerol -O- acyltransferase 1), leptin C528T und TG 5’
UTR (thyroglobulin). Aus den Blut- und Haareproben wurden die Genotypisierungen für DGAT1 K232A und Leptin C528T Polymorphismus mit TaqMan (allelic discrimination methode) in einer Rotor-Gene RG 3000 Real-Time PCR-System benutzt, für den TG 5’
UTR Polymorphismus wurden die Auswertungen mit PCR-RFLP Methode durchgeführt.
In der Bestimmung sowohl Real-Time PCR-System als auch PCR-RFLP Methoden wurden für alle Locus drei verschiedenen Allele isoliert (DGAT1: AA/AA, AA/GC, GC/GC; leptin: CC, CT, TT; TG: CC, CT, TT).
Nach den Genotypisierungen wurden die Berechnungen von Allelfrequenzen und populationsgenetischen Parametern ausgewertet, und wurden die Zusammenhänge zwischen den Genotypen und Milch- und Fleischproduktionsparametern mit den Programmpaketen SPSS 14.0 statistisch kontrolliert.
Es wurde signifikante Zusammenhang zwischen den Genotypen (DGAT1 K232A, leptin C528T und TG 5’ UTR) der drei Ungarische Milchkühepopulationen (Ungarisches Fleckvieh, Holstein Friesian, Jersey) und denen Milchproduktionseigenschaften gefunden.
Die Autorin fand signifikante Zusammenhang zwischen den Genotypen (DGAT1 K232A, leptin C528T und TG 5’ UTR) den Angus Stieren und den Fettinhälte (%) in dem Musculus longissimus dorsi (LD) und Musculus semitendinosus (ST).
1. TARTALOMJEGYZÉK
1. TARTALOMJEGYZÉK... 8
2. RÖVIDÍTÉSEK JEGYZÉKE ... 11
3. TÁBLÁZATOK JEGYTÉKE ... 13
4. ÁBRAJEGYZÉK ... 16
5. BEVEZETÉS ... 18
6. IRODALMI ÁTTEKINTÉS ... 20
6.1. A mennyiségi tulajdonságok elemzése ... 20
6.1.1. A genetikai markerek az állattenyésztésben... 22
6.1.2. Polimorfizmusok, és kimutatásuk eszközei ... 24
6.1.3. A valós idejű polimeráz láncreakció jellemzése ... 30
6.1.4. A PCR-RFLP módszer jellemzése ... 31
6.2. A szarvasmarha genetika jelentősége ... 31
6.3. A gén felépítése és működése ... 32
6.4. Diacilglicerol -O- aciltranszferáz (DGAT1) mint marker gén ... 35
6.4.1. A diacilglicerol -O- aciltranszferáz (DGAT1) ... 35
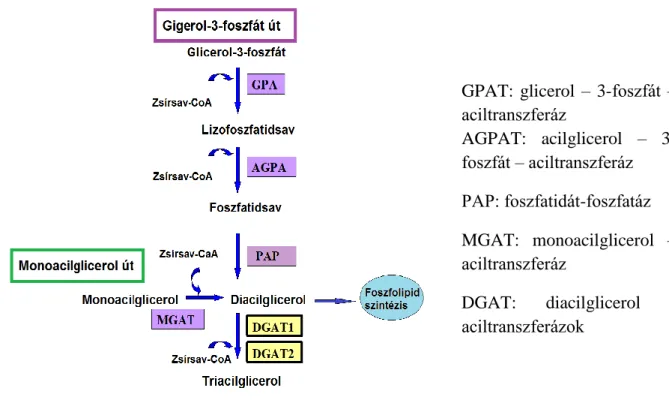
6.4.2. A trigliceridek szintézise ... 35
6.4.2.1. A glicerol-3-foszfát út ... 36
6.4.2.2. A monoacilglicerol út ... 36
6.4.3. Zsír szintézis a tőgy mirigyekben... 37
6.4.4. DGAT1 és DGAT2 gén családok... 37
6.4.5. A DGAT1 gén K 232 A polimorfizmusa ... 38
6.5. Leptin mint marker gén ... 40
6.5.1. A leptin ... 40
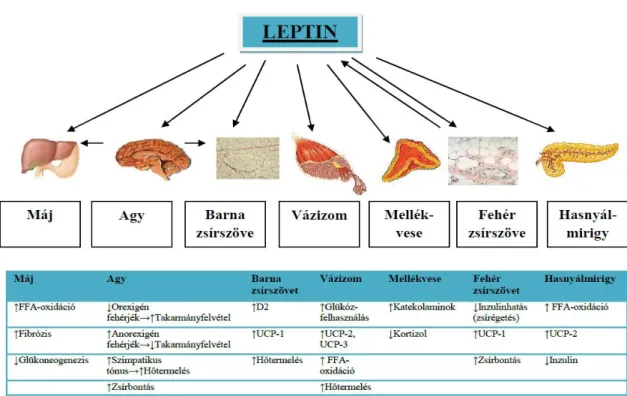
6.5.2. A leptin szerepe, hatásai ... 40
6.5.3. A pajzsmirigyhormonok és a leptin ... 43
6.5.6. A leptin C528T polimorfizmusa ... 43
6.6. Thyroglobulin (TG) mint marker gén ... 44
6.6.1. A thyroglobulin (TG) ... 44
6.6.2. A pajzsmirigyhormonok és szintézisük... 44
6.6.3. A pajzsmirigyhormonok szerepe, hatásai... 46
6.6.4. A TG 5’ UTR polimorfizmusa ... 47
7. A VIZSGÁLAT CÉLJA ... 48
8. ANYAG ÉS MÓDSZER ... 49
8.1. A vizsgálatba vont fajták, egyedszámok ... 49
8.2. Mintaelőkészítés ... 51
8.2.1. Mintavétel... 51
8.2.2. A genomiális DNS izolálása ... 51
8.2.3. A hosszú hátizom (LD) és a fehérpecsenye (ST) zsírtartalmának meghatározása Soxhlet féle gravimetriás módszerrel ... 52
8.3. A DGAT1 gén K232A és a leptin C528T polimorfizmusának vizsgálata Real-time PCR, TaqMan allélikus diszkrimináció módszerével ... 53
8.3.1. A Real-Time PCR reakció... 53
8.3.1.1. A DGAT1 gén K232A polimorfizmusának vizsgálata Real-time PCR, TaqMan allélikus diszkrimináció módszerével ... 54
8.3.1.2. A leptin C528T polimorfizmusának vizsgálata Real-time PCR, TaqMan allélikus diszkrimináció módszerével ... 55
8.4. A TG gén 5’UTR-polimorfizmusának vizsgálata PCR-RFLP módszerrel ... 56
8.4.1. A polimeráz láncreakció... 56
8.4.2. Restrikciós emésztés (RFLP) ... 57
8.4.3. Az elektroforézis ... 57
8.5. Adatbáziskezelés és statisztikai vizsgálatok ... 58
8.5.1. Statisztikai analízis ... 58
8.5.2.1. Populációegyensúly-vizsgálatok ... 58
8.5.2.2. Hatásvizsgálat ... 60
8.5.2.2.1. Általános Lineáris Modell (GLM) ... 60
8.5.2.2.2. Dominancia és additív hatás ... 61
9. EREDMÉNYEK ÉS ÉRTÉKELÉSÜK ... 62
9.1. Allél- és genotípus-megoszlási értékek ... 62
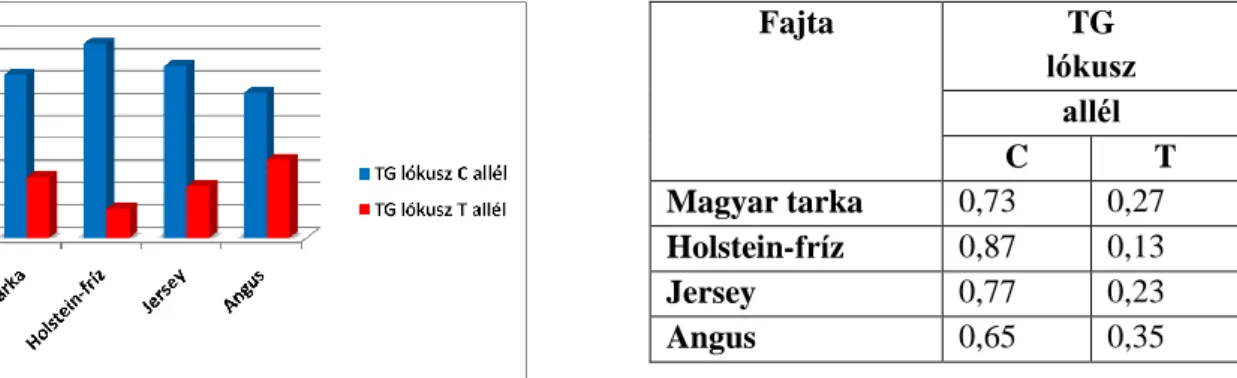
9.1.1. A DGAT1 gén K232A polimorfizmusának genotípus- és allélgyakorisági értékei hazai szarvasmarha populációkban ... 62
9.1.2. A leptin gén C528T polimorfizmusának genotípus- és allélgyakorisági értékei hazai szarvasmarha populációkban ... 67
9.1.3. A TG gén 5’UTR polimorfizmusának genotípus- és allélgyakorisági értékei hazai szarvasmarha populációkban ... 70
9.2. Statisztikai analízis eredményei ... 74
9.2.1. Leíró statisztika eredményei... 74
9.2.2. Hatásvizsgálat eredményei ... 78
9.2.2.1. A DGAT1 K232A, a leptin C528T, valamint a TG 5’ UTR polimorfizmusok és a vizsgált tulajdonságok közötti összefüggések hazai magyar tarka állományban... 78
9.2.2.1.1. Többváltozós varianciaanalízis eredményeinek összehasonlítása ... 78
9.2.2.1.2. A független változók hatásai ... 81
9.2.2.1.3. A genotípus-átlagok összevetése ... 82
9.2.2.2. A DGAT1 K232A, a leptin C528T, valamint a TG 5’ UTR polimorfizmusok és a vizsgált tulajdonságok közötti összefüggések hazai holstein- fríz állományban ... 94
9.2.2.2.1. Többváltozós varianciaanalízis eredményeinek összehasonlítása ... 94
9.2.2.2.2. A független változók hatásai ... 96
9.2.2.2.3. A genotípus-átlagok összevetése ... 97
9.2.2.3. A DGAT1 K232A, a leptin C528T, valamint a TG 5’ UTR polimorfizmusok és a vizsgált tulajdonságok közötti összefüggések hazai jersey állományban ... 106
9.2.2.3.1. Többváltozós varianciaanalízis eredményeinek összehasonlítása ... 106
9.2.2.3.2. A független változók hatásai ... 107
9.2.2.3.3. A genotípus-átlagok összevetése ... 108
9.2.2.4. A DGAT1 K232A, a leptin C528T valamint a TG 5’ UTR polimorfizmusok
és a vizsgált tulajdonságok közötti összefüggések hazai angus állományban... 116
9.2.2.4.1. Többváltozós varianciaanalízis eredményeinek összehasonlítása ... 116
9.2.2.4.2. A független változók hatásai ... 117
9.2.2.4.3. A genotípus-átlagok összevetése ... 118
9.2.2.5. A vizsgált tejelő magyar tarka, holstein-fríz és jersey populációk összehasonlítása 305 napos laktációs termelési mutatóik valamint különböző genotípusaik alapján. ... 123
10. KÖVETKEZTETÉSEK ÉS JAVASLATOK ... 125
11. ÚJ KUTATÁSI EREDMÉNYEK (TÉZISPONTOK) ... 127
12. NEW RESEARCH RESULTS (POINTS OF THESIS) ... 129
13. ÖSSZEFOGLALÁS ... 131
14. KÖSZÖNETNYILVÁNÍTÁS ... 137
15. TUDOMÁNYOS KÖZLEMÉNYEK JEGYZÉKE ... 138
15.1. Az értekezés témakörében megjelent tudományos közlemények ... 138
15.1.1. Idegen nyelvű folyóiratban megjelent lektorált cikk ... 138
15.1.2. Magyar nyelvű folyóiratban megjelent lektorált cikk ... 138
15.1.3. Konferencia kiadványban megjelent idegen nyelvű közlemények ... 138
15.1.4. Konferencia kiadványban megjelent magyar nyelvű közlemények ... 139
15.2. Értekezés témakörén kívül (egyéb) közlemények ... 139
15.2.1. Idegen nyelvű folyóiratban megjelent lektorált cikk ... 139
15.2.2. Magyar nyelvű folyóiratban megjelent lektorált cikk ... 140
15.2.3. Konferencia kiadványban megjelent magyar nyelvű közlemények ... 140
16. FELHASZNÁLT IRODALOM ... 141
17. MELLÉKLETEK ... 156
2. RÖVIDÍTÉSEK JEGYZÉKE
5’D: 5’-dejodáció 5D: 5-dejodáció A: adenin
ACAT1, ACAT2: acil-CoA: koleszterin acil-transzferázok
AFLP: Amplified Fragment Length Polymorphism - amplifikált fragmenthossz polimorfizmus
AGPAT: acilglicerol – 3-foszfát – aciltranszferáz
AP-PCR: Arbitrary Primered PCR - PCR tetszés szerinti primerrel ARMS: Amplification Refractory Mutation System
ASA: Allele Specific Amplification – allél specifikus amplifikáció ASP: Allele Specific PCR– allél specifikus polimeráz láncreakció bp: bázispár
C: citozin
cM: centimorgan D1: I-es típusú dejodáz D2: II-es típusú dejodáz D3: III-as típusú dejodáz
DAF: (DNA Amplification Fingerprinting = DNS amplifikációs ujjlenyomat) DGAT: diacilglicerol - aciltranszferázok
DGAT1: diacilglicerol-O-acyltranszferáz
DOP-PCR: degenerált oligonukleotid által vezetett amplifikáció EC: extracelluláris domén
FFA: Free fatty acid – szabad zsírsav fT3: szabad állapotú trijód-tironin fT4: szabad állapotú tiroxin FZS: fehér zsírszövet G: guanin
GH: Growth hormone – növekedési hormon
GLM: General linear model - általános lineáris modell
GMS: Genomic Mismatch Scanning > azonosságok kihasználása GPAT: glicerol – 3-foszfát – aciltranszferáz
GS: genomszelekció IC: intracelluláris domén
IGF-1: Insulin-like growth factor – inzulinszerű növekedési faktor K: lizin
LCR: Ligase Chain Reaction – ligáz láncreakció
MAS: Marker Assisted Selection – markerekre alapozott szelekció MGAT: monoacilglicerol – aciltranszferáz
MOPAC: kevert oligonukleotid vezetett cDNS amlifikáció mRNS: messenger RNS – hírvivő ribonukleinsav
Ob: „obesitas”- elhízás Ob-R: Leptin receptor PAP: foszfatidát-foszfatáz
PASA: PCR Amplification of Specific Alleles
PCR: Polymerase Chain Reaction – polimeráz láncreakció PEP: primer extenziós előamplifikáció
PM: Pajzsmirigy
PMH: Pajzsmirigyhormon
QTL: Quantitative Trait Locus – értékmérő tulajdonságok génhelye
RAPD: Random Amplified Polimorphic DNA = random amplifikált polimorf DNS) RDA: Representational Difference Analysis > különbségek felderítése
RFLP: Restriction Fragment Length Polymorphism - restrikciós fragmenthossz- polimorfizmus
rT3: reverz trijód-tironin
SNP: Single Nucleotide Polymorphism, („sznip”) – egypontos nukleotid-polimorfizmus SSCP: Single-Strand Conformation Polymorphism – egyszálú DNS konfirmációs polimorfizmus
SSR: Simple Sequence Repeat – egyszerű szekvencia ismétlődés STR: Short Tandem Repeat – rövid tandem ismétlődés
T: timin
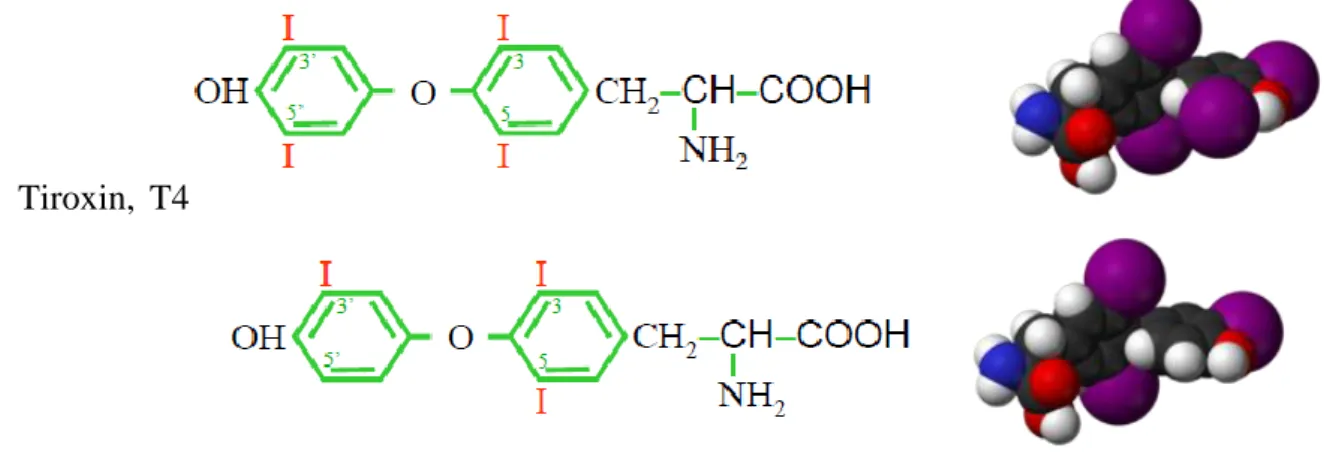
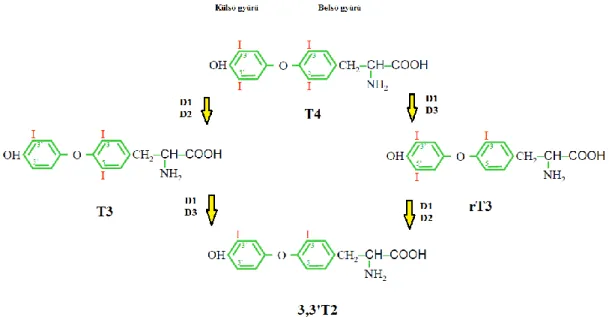
T2: dijód-tironin T3: trijód-tironin T4: tiroxin
TG: tieroglobulin vagy thyroglobulin
TR: Thyroid receptor – pajzsmirigyhormon-receptor
TRH: Thyreotrop releasing hormone – tireotrop elválasztást serkentő hormon TSH: Thyroid stimulating hormone – pajzsmirigy-serkentő hormon
UCP: Uncoupling protein - szétkapcsoló fehérje UTR: Untranslated region – nem transzlálódó régió
VNTR: Variable Number of Tandem Repeats – eltérő számú tandem ismétlődések
3. TÁBLÁZATOK JEGYTÉKE
1. táblázat: A szarvasmarha fontosabb értékmérő tulajdonság csoportjaiban felfedezett QTL-ek száma ... 21 2. táblázat: Polimorfizmusok kimutatásához használt eljárások... 27 3. táblázat: A vizsgált szarvasmarha populáció tenyészetenkénti megoszlása, elemszámokkal ... 49 4. táblázat: A DGAT1 gén K232A genotípusok megoszlása és 2 értékei a vizsgált hazai fajtákban. ... 64 5. táblázat: A DGAT1 gén K232A allélgyakoriságok a vizsgált hazai fajtákban. ... 64 6. táblázat: A DGAT1 K232A polimorfizmus megoszlása a különböző szarvasmarha fajtákban. ... 65 7. táblázat: A leptin gén C528T genotípusok megoszlása és 2 értékei a vizsgált hazai fajtákban. ... 68 8. táblázat: A leptin gén 258 (C/T) allélgyakoriságok a vizsgált hazai fajtákban. ... 68 9. táblázat: A leptin C528T polimorfizmus megoszlása a különböző szarvasmarha fajtákban. ... 69 10. táblázat: A TG 5’ genotípusok megoszlása és 2 értékei a vizsgált hazai fajtákban. ... 71 11. táblázat: A TG gén 5’ allélgyakoriságok a vizsgált hazai fajtákban. ... 72 12. táblázat: A TG 5’ polimorfizmus megoszlása a különböző szarvasmarha fajtákban. .. 72 13. táblázat: A vizsgált magyar tarka tehén populáció laktációs tulajdonságainak leíró statisztikája az egyes DGAT1, leptin és TG genotípusok szerinti bontásban. ... 75 14. táblázat: A vizsgált holstein-fríz tehén populáció laktációs tulajdonságainak leíró statisztikája az egyes DGAT1, leptin és TG genotípusok szerinti bontásban. ... 76 15. táblázat: A vizsgált jersey tehén populáció laktációs tulajdonságainak leíró statisztikája az egyes DGAT1, leptin és TG genotípusok szerinti bontásban. ... 77 16. táblázat: A vizsgált angus populáció hosszú hátizom- és fehérpecsenye zsírtartalmának leíró statisztikája az egyes DGAT1, leptin és TG genotípusok szerinti bontásban. ... 78 17. táblázat. A magyar tarka populáció esetében alkalmazott modellben használt faktorok tejtermelési tulajdonságokra gyakorolt hatásai többváltozós varianciaanalízis módszerével számolva (U-próba). ... 79 18. táblázat: A továbbfejlesztett modellben használt faktorok tejtermelési tulajdonságokra gyakorolt hatásai többváltozós varianciaanalízis módszerével számolva, magyar tarka populáció esetében (U-próba). ... 80 19. táblázat: A vizsgált hatótényezők hatásai, illetve a hatások erőssége (szignifikancia szintje) tulajdonságok szerinti bontásban a magyar tarka populáció esetében. ... 82 20. táblázat: A varianciák homogenitását vizsgáló Levene-féle teszt eredményei a tejtermelési mutatóknál... 83 21. táblázat: A tejtermelési mutatók legkisebb négyzetes átlagai és azok hibái DGAT1, leptin és TG genotípusonként hazai magyar tarka populáció esetében. ... 83 20. ábra: A maximálisan illetve az átlagosan befejt tejmennyiség legkisebb négyzetes átlagai DGAT1, genotípusonként. ... 84
21. ábra: A maximálisan illetve az átlagosan befejt tejmennyiség legkisebb négyzetes átlagai leptin genotípusonként. ... 84 22. ábra: A maximálisan illetve az átlagosan befejt tejmennyiség legkisebb négyzetes átlagai TG genotípusonként. ... 85 22. táblázat: A 305 napos laktációs termelési mutatók legkisebb négyzetes átlagai és azok hibái DGAT1, leptin és TG genotípusonként hazai magyar tarka populáció esetében. ... 86 23. táblázat: A vizsgált hazai magyar tarka populáció 305 napos laktációs termelési mutatóinak legkisebb négyzetes átlagai, valamint az additív hatás, dominancia és variancia értékei a DGAT1 genotípus esetében. ... 91 24. táblázat: A vizsgált hazai magyar tarka populáció 305 napos laktációs termelési mutatóinak legkisebb négyzetes átlagai, valamint az additív hatás, dominancia és variancia értékei a leptin genotípus esetében. ... 92 25. táblázat: A vizsgált hazai magyar tarka populáció 305 napos laktációs termelési mutatóinak legkisebb négyzetes átlagai, valamint az additív hatás, dominancia és variancia értékei a TG genotípus esetében. ... 93 26. táblázat: A modellben használt faktorok tejtermelési tulajdonságokra gyakorolt hatásai többváltozós varianciaanalízis módszerével számolva (U-próba) hazai holstein-fríz populáció esetében. ... 94 27. táblázat: A továbbfejlesztett modellben használt faktorok tejtermelési tulajdonságokra gyakorolt hatásai többváltozós varianciaanalízis módszerével számolva, holstein-fríz populáció esetében (U-próba). ... 95 28. táblázat: A vizsgált faktorok hatásai, illetve a hatások erőssége (szignifikancia szintje) tulajdonságok szerinti bontásban a holstein-fríz populáció esetében. ... 96 29. táblázat: A varianciák homogenitását vizsgáló Levene-féle teszt eredményei a tejtermelési mutatóknál... 97 30. táblázat: A tejtermelési mutatók legkisebb négyzetes átlagai és azok hibái DGAT1, leptin és TG genotípusonként hazai holstein-fríz populáció esetében. ... 98 31. táblázat: A 305 napos laktációs termelési mutatók legkisebb négyzetes átlagai és azok hibái DGAT1, leptin és TG genotípusonként hazai holstein-fríz populáció esetében. ... 99 32. táblázat: A vizsgált hazai holstein-fríz állomány 305 napos laktációs termelési mutatók legkisebb négyzetes átlagai, valamint az additív hatás, dominancia és variancia értékei a DGAT1 genotípus esetében. ... 104 33. táblázat: A vizsgált hazai holstein-fríz állomány 305 napos laktációs termelési mutatók legkisebb négyzetes átlagai, valamint az additív hatás, dominancia és variancia értékei a leptin genotípus esetében. ... 104 34. táblázat: A vizsgált hazai holstein-fríz populáció 305 napos laktációs termelési mutatók legkisebb négyzetes átlagai, valamint az additív hatás, dominancia és variancia értékei a TG genotípus esetében. ... 105 35. táblázat: A modellben használt faktorok tejtermelési tulajdonságokra gyakorolt hatásai többváltozós varianciaanalízis módszerével számolva (U-próba) hazai jersey populáció esetében. ... 106 36. táblázat: A vizsgált hatótényezők hatásai, illetve a hatások erőssége (szignifikancia szintje) tulajdonságok szerinti bontásban jersey populáció esetében. ... 108 37. táblázat: A varianciák homogenitását vizsgáló Levene-féle teszt eredményei a tejtermelési mutatóknál... 109
38. táblázat: A tejtermelési mutatók legkisebb négyzetes átlagai és azok hibái DGAT1, leptin és TG genotípusonként hazai jersey populáció esetében. ... 109 39. táblázat: A 305 napos laktációs termelési mutatók legkisebb négyzetes átlagai és azok hibái DGAT1, leptin és TG genotípusonként hazai jersey populáció esetében. ... 110 40. táblázat: A vizsgált jersey populáció 305 napos laktációs termelési mutatók legkisebb négyzetes átlagai, valamint az additív hatás, dominancia és variancia értékei a DGAT1 genotípus esetében. ... 114 41. táblázat: A vizsgált jersey populáció 305 napos laktációs termelési mutatók legkisebb négyzetes átlagai, valamint az additív hatás, dominancia és variancia értékei a leptin genotípus esetében. ... 115 43. táblázat: A modellben használt faktorok tejtermelési tulajdonságokra gyakorolt hatásai többváltozós varianciaanalízis módszerével számolva (U-próba) az angus populáció esetében. ... 116 44. táblázat: A vizsgált hatótényezők hatásai, illetve a hatások erőssége (szignifikancia szintje) tulajdonságok szerinti bontásban angus populáció esetében. ... 117 45. táblázat: A varianciák homogenitását vizsgáló Levene-féle teszt eredményei. ... 118 46. táblázat: A vizsgált hosszú hátizom (m. longissimus dorsi) és fehérpecsenye (m.
semitendinosus) zsírtartalmának legkisebb négyzetes átlagai és azok hibái DGAT1, leptin és TG genotípusonként az angus populáció esetében. ... 118 47. táblázat: A vizsgált angus populáció hosszú hátizom (m. longissimus dorsi) és fehérpecsenye (m. semitendinosus) zsírtartalmának legkisebb négyzetes átlagai, valamint az additív hatás, a dominancia és a variancia értékei a DGAT1 genotípus esetében. ... 121 48. táblázat: A vizsgált angus populáció hosszú hátizom (m. longissimus dorsi) és fehérpecsenye (m. semitendinosus) zsírtartalmának legkisebb négyzetes átlagai, valamint az additív hatás, a dominancia és a variancia értékei a leptin genotípus esetében. ... 122 49. táblázat: A vizsgált angus populáció hosszú hátizom (m. longissimus dorsi) és fehérpecsenye (m. semitendinosus) zsírtartalmának legkisebb négyzetes átlagai, valamint az additív hatás, a dominancia és a variancia értékei a TG genotípus esetében. ... 122 50. táblázat: A 305 napos laktációs termelési mutatók legkisebb négyzetes átlagai és azok hibái DGAT1, genotípusonként a vizsgált tejelő magyar tarka, holstein-fríz és jersey populációkban. ... 123 51. táblázat: A 305 napos laktációs termelési mutatók legkisebb négyzetes átlagai és azok hibái leptin genotípusonként a vizsgált tejelő magyar tarka, holstein-fríz és jersey populációkban. ... 124 52. táblázat: A 305 napos laktációs termelési mutatók legkisebb négyzetes átlagai és azok hibái TG genotípusonként a vizsgált tejelő magyar tarka, holstein-fríz és jersey populációkban. ... 124
4. ÁBRAJEGYZÉK
1. ábra: A QTL-ek meghatározásának sematikus ábrája ... 20
2. ábra: Hosszúságpolimorfizmus és egypontos nukleotid variáció. ... 24
3. ábra: SNP: Egypontos nukleotid-polimorfizmus. ... 25
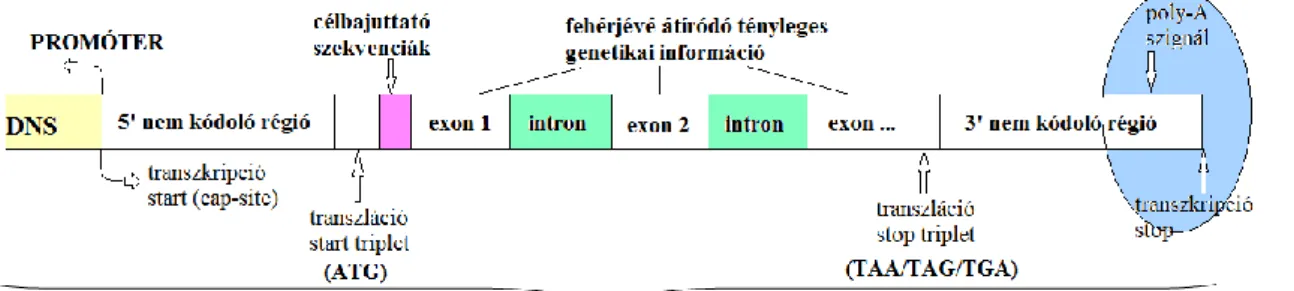
4. ábra: A gén három fő egysége. ... 32
5. ábra: A promóter felépítése. ... 33
6. ábra: Az eukariota gén szerkezete . ... 34
7. ábra: A DGAT1 enzim szerkezete ... 35
8. ábra: A trigliceridek szintézise ... 36
9. ábra: A DGAT1, DGAT2 enzimek szerepét szemléltető ábra a triglicerid szintézis folyamán az endoplazmatikus retikulumban. ... 38
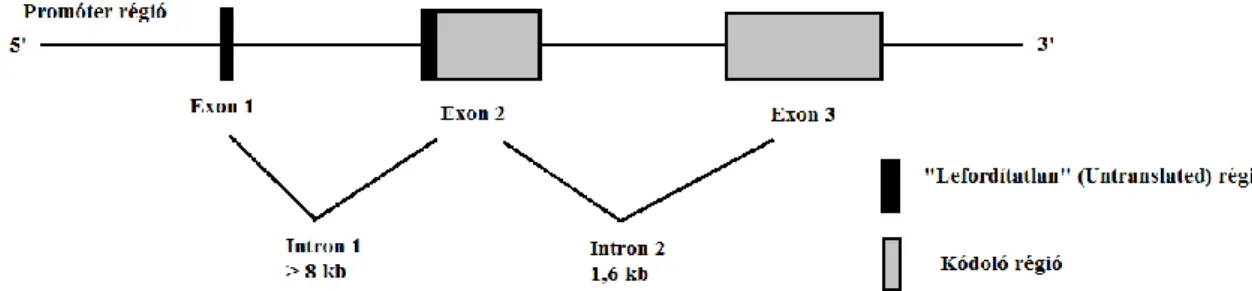
10. ábra: A szarvasmarha DGAT1 gén exon, intron struktúrája. ... 39
11. ábra: A leptin centrális és perifériás hatásai. ... 41
12. ábra: A különféle leptin receptor izoformák. ... 42
13. ábra: A leptin gén exon, intron struktúrája. ... 43
14. ábra: A pajzsmitigyhormonok felépítése. ... 44
15. ábra: A pajzsmirigyhormonok átalakulása a dejodáció során. ... 45
16. ábra: A DGAT1 genotípusok kiértékelése Rotor Gene Analysis 6.1 szoftver segítségével. ... 62
17. ábra: A DGAT1 gén K232A polimorfizmus változatai. ... 63
18. ábra: A leptin gén C528T polimorfizmus változatai. ... 67
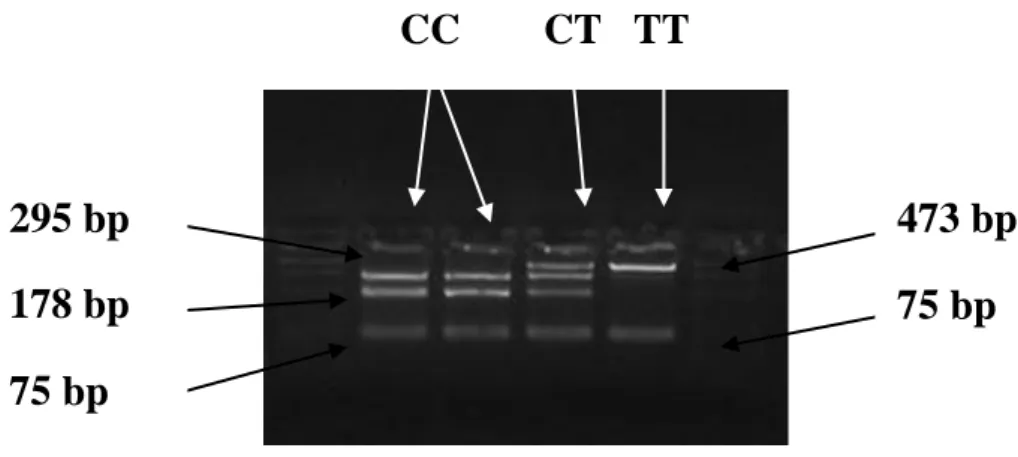
19. ábra: A szarvasmarha TG gén 5’ változatai 4%-os agaróz gélen. ... 70
20. ábra: A maximálisan illetve az átlagosan befejt tejmennyiség legkisebb négyzetes átlagai DGAT1, genotípusonként. ... 84
21. ábra: A maximálisan illetve az átlagosan befejt tejmennyiség legkisebb négyzetes átlagai leptin genotípusonként. ... 84
22. ábra: A maximálisan illetve az átlagosan befejt tejmennyiség legkisebb négyzetes átlagai TG genotípusonként. ... 85
23. ábra: A 305 napos laktációs tejhozam legkisebb négyzetes átlagai DGAT1 genotípusonként. ... 86
24. ábra: A 305 napos laktációs tejhozam legkisebb négyzetes átlagai leptin genotípusonként. ... 87
25. ábra: A 305 napos laktációs tejhozam legkisebb négyzetes átlagai TG genotípusonként. ... 87
26. ábra: A 305 napos laktációs tejzsír és fehérje százalék legkisebb négyzetes átlagai DGAT1 genotípusonként. ... 88
27. ábra: A 305 napos laktációs tejzsír és fehérje százalék legkisebb négyzetes átlagai leptin genotípusonként. ... 88
28. ábra: A 305 napos laktációs tejzsír és fehérje százalék legkisebb négyzetes átlagai TG genotípusonként. ... 89 29. ábra: A 305 napos laktációs tejhozam legkisebb négyzetes átlagai DGAT1
30. ábra: A 305 napos laktációs tejhozam legkisebb négyzetes átlagai leptin
genotípusonként. ... 100 31. ábra: A 305 napos laktációs tejhozam legkisebb négyzetes átlagai TG
genotípusonként. ... 100 32. ábra: A 305 napos laktációs tejzsír, és fehérje százalék legkisebb négyzetes átlagai DGAT1 genotípusonként. ... 101 33. ábra: A 305 napos laktációs tejzsír, és fehérje százalék legkisebb négyzetes átlagai leptin genotípusonként. ... 102 34. ábra: A 305 napos laktációs tejzsír, és fehérje százalék legkisebb négyzetes átlagai TG genotípusonként. ... 102 35. ábra: A 305 napos laktációs tejhozam legkisebb négyzetes átlagai DGAT1
genotípusonként. ... 111 36. ábra: A 305 napos laktációs tejhozam legkisebb négyzetes átlagai leptin
genotípusonként. ... 111 37. ábra: A 305 napos laktációs tejhozam legkisebb négyzetes átlagai TG
genotípusonként. ... 111 38. ábra: A 305 napos laktációs tejzsír, és fehérje százalék legkisebb négyzetes átlagai DGAT1 genotípusonként. ... 112 39. ábra: A 305 napos laktációs tejzsír, és fehérje százalék legkisebb négyzetes átlagai leptin genotípusonként. ... 113 40. ábra: A 305 napos laktációs tejzsír, és fehérje százalék legkisebb négyzetes átlagai TG genotípusonként. ... 113 41. ábra: A hosszú hátizom (m. longissimus dorsi) és fehérpecsenye (m. semitendinosus) zsírtartalmának legkisebb négyzetes átlagai DGAT1 genotípusonként. ... 119 42. ábra: A hosszú hátizom (m. longissimus dorsi) és fehérpecsenye (m. semitendinosus) zsírtartalmának legkisebb négyzetes átlagai leptin genotípusonként. ... 119 43. ábra: A hosszú hátizom (m. longissimus dorsi) és fehérpecsenye (m. semitendinosus) zsírtartalmának legkisebb négyzetes átlagai TG genotípusonként. ... 120
5. BEVEZETÉS
Jelenleg 7 milliárd ember él a Földön, s miközben világszerte csökken a termékenységi ráta, becslések szerint a Föld lakossága 2050-re elérheti a 9 milliárdot. Az OECD-FAO jóslatai szerint az előttünk álló 10 évben az élelmiszerfogyasztás meg fog duplázódni a világon (1. melléklet). Ez a változás új kihívást jelent az élelmiszertermelés, ezen belül az állattenyésztés területén. A helyzet megoldásául szolgálhat, hogy a tenyésztők a több és jobb minőségű állati eredetű élelmiszereket nem az állatlétszám növelésével, hanem elsősorban a termelés hatékonyságának fokozásával próbálják elérni.
A biológiai, genetikai ismeretek és módszerek egyre szélesebb körű megismerése újabb lehetőségeket kínálnak a szelekció hatékonyságának a növelésére. A hatékonyság növelését elsősorban az állatállomány genetikai képességének javítása, a minél pontosabb tenyészértékbecslés és a hatékony szelekció alapozhatja meg.
Az új biotechnológiai eljárások alkalmazásának legnagyobb haszonélvezője a mezőgazdaság és az élelmiszeripar. Ezekre az ágazatokra jut a biotechnológiai tevékenységek bővülésének 70%-a. Így bátran kijelenthetjük, hogy a biotechnológia kutatási eredményeire alapozott technológiák a piaci versenyképességet és a gazdaságosságot nagymértékben meghatározó tényezővé válnak a világgazdaságban. A biotechnológiai eljárások sok olyan gyakorlati probléma megoldására kínálnak lehetőséget, amelyekre régebben nem is gondolhattunk.
Mára képesek lettünk genetikai vizsgálatok útján, közvetlenül a DNS szekvencia alapján megállapítani azt, hogy egy bizonyos egyed hordoz-e egy adott tulajdonságot vagy sem. Gazdasági jelentősége miatt a szarvasmarha volt az egyik első olyan háziállat, melynek meghatározták a genomszekvenciáját. Az állattenyésztők számára fontos értékmérő tulajdonságok többségét (tej-, hús-, tojás-, gyapjútermelés) több gén együttes hatása, valamint a környezet alakítja ki. Az eddigi kutatásokban számos QTL-t (a quantitative trait locus vagy QTL az a régió a kromoszómákon belül, ahol a mennyiségi tulajdonság kialakításában szerepet játszó gének helyezkednek el.) azonosítottak főleg tejelő szarvasmarha fajtákban. Jelenleg a kutatók fő célja az egyes QTL-ek mögött rejlő mutációkat hordozó gének azonosítása, ami sokkal hatékonyabbá fogja tenni a marker alapú szelekciót (Marker assisted selection: MAS). A markerekre alapozott szelekció gyakorlati alkalmazásával a fajták előállítási ideje lerövidíthető, ezáltal gazdaságosabbá tehető. Mindezek előfeltétele azonban az adott tulajdonsághoz szorosan kapcsolt markerek detektálása. A számos ismert marker között jelentősek a mennyiségi tulajdonság kialakításában szerepet játszó diacilglicerol-O-acyltranszferáz (DGAT1), tireoglobulin (TG) és a leptin, amelyek egyre inkább a vizsgálatok középpontjába kerültek, és vizsgálataim alapját képezték.
A gazdasági állatfajok közül a szarvasmarha-tenyésztés hazánkban és világviszonylatban is igen nagy jelentőségű ágazata a mezőgazdaságnak. Magyarországon a szarvasmarhák száma több évtizedes mérséklődést követően a 2000-es évek végén stabilizálódott, 2010-ben azonban tovább csökkent. 2010 decemberében a gazdaságok 681 ezer szarvasmarhát tartottak. A tehénállomány 309 ezer darab volt, nem csökkent
jelentősen. A tejhasznú tehenek száma 19 ezerrel mérséklődött, a húshasznú tehénállomány viszont 6 ezerrel lett több. Az állati termékek közül tehéntejből 1,6 milliárd litert, az előző évinél 7%-kal kevesebbet termeltek a gazdaságok (Központi Statisztikai Hivatal: KSH, Mezőgazdaság 2010). A világ tejtermelése és az ehhez szorosan kapcsolódó tejfeldolgozás Európában és Észak-Amerikában koncentrálódik. A világon megtermelt tej mintegy fele Európából származik és a világ összes tejtermelésének közel háromnegyedét adja együttesen Európa és Észak-Amerika.
A szarvasmarha-tenyésztés az elsősorban emberi táplálékként hasznosított tej- és hústermelés mellett fontos ipari nyersanyagokat is előállít. A tej és tejtermékek a legértékesebb élelmiszereink közé tartozik. A legújabb kutatási eredményeket összegezve egyértelműen megállapítható, hogy az összes élelmiszer közül a tej és a belőle készített termékek a leggazdagabbak a bioaktív anyagokban. Ez a megállapítás a tej valamennyi makro- és mikro alkotórész-csoportjára vonatkozik, közte a sokat támadott tejzsírra is. A tej ipari feldolgozási arányának növekedésével, a termékek minőségével szembeni követelmények előtérbe kerülésével, a tej alkotórészek fontos szelekciós követelményként szerepelnek. Bár jelenleg ármeghatározó szerepük nem jellemző, látva a fejlettebb tej- és tejtermék fogyasztási kultúrával rendelkező országok árképzését, várhatóan a közeljövő tejárképzési rendszerében a tejzsír és a tejfehérje, valamint ezek összetevői a korábbiaknál meghatározóbb szerepet fognak játszani.
A rohamléptekkel fejlődő molekuláris genetikai módszerek közül az SNP (single nucleotide polymorphism - egypontos nukleotid-polimorfizmus) kutatások (köztük a DGAT1, leptin, TG) új lehetőséget kínálnak a genomvizsgálatokon alapuló tenyészállat kiválasztásban, azaz a genomszelekcióban (GS). Ami azért is fontos, mert a genom alapú tenyészérték felhasználásával nagyobb mértékű genetikai előrehaladás érhető el.
Az általam vizsgált polimorfizmusokat (DGAT1 K232A, TG 5’ UTR, leptin C528T), mint fontos mennyiségi tulajdonságokat meghatározó marker géneket, már számos külföldi országban sikeresen alkalmazzák a hatékonyabb szelekciós munka részeként.
Munkám során arra igyekeztem választ kapni, hogy ezek a marker gének miként befolyásolják a tej termelést, a tej összetételét, valamint a marhahús faggyú tartalmát.
Dolgozatom célja tehát, hogy a szarvasmarha DGAT1 K232A, leptin C528T és TG 5’ UTR polimorfizmusokat, mint a tejtermelés és az intramuszkuláris zsírtartalom lehetséges genetikai markereit vizsgálva kapcsolatot keressek az előbb említett tulajdonságok és az enzim genotípusok között.
6. IRODALMI ÁTTEKINTÉS
6.1. A mennyiségi tulajdonságok elemzése
Az állattenyésztésben a gazdaságilag jelentős értékmérő tulajdonságok többsége mennyiségi (kvantitatív) jellegű. Ezek általában additív módon öröklődnek. A megfigyelések folyamatos, normális eloszlással jellemezhetők és a szokásos mértékegységekkel kifejezhetők. Ennek oka, hogy a fenotípust a genotípus és a környezet nagymértékben befolyásolja, és a genotípust nagyszámú gén alakítja ki. Ide tartoznak pl. a szaporaság, a tejtermelés, a takarmányértékesítés, gyapjúhozam, vágási százalék, életteljesítmény. Mivel a mennyiségi tulajdonságok kialakulásában több száz vagy ezer gén is szerepet játszhat, ezért ezeknek az elemzése lényegesen nehezebb, mint a kvalitatív tulajdonságoké.
1. ábra: A QTL-ek meghatározásának sematikus ábrája (www.animalgenom.org nyomán Farkas). [A mennyiségi tulajdonság kialakításában szerepet játszó gének hatásának elemzésére és a kromoszómán elfoglalt helyük meghatározására alkalmazott térképezést követő feladat a szakaszokon található gének azonosítása. A QTL-ek szekvenálásával lehetőség nyílik az összehasonlításra már ismert génekkel is.]
Mint az már a bevezetésben szóba került, a QTL az a régió a kromoszómákon belül, ahol a mennyiségi tulajdonság kialakításában szerepet játszó gének helyezkednek el. Ha QTL-ről beszélünk, akkor ez alatt a kromoszóma egy viszonylag szűk régióját értjük, mely azonban több gént is magában foglalhat. Egy adott jelleg kialakításában általában egy, vagy néhány nagy (major), és több, változó számú kis (minor) gén vesz részt.
A markertechnika új lehetőséget nyitott meg a gének hatásának elemzésére és a kromoszómán elfoglalt helyük meghatározására. Nagy előrelépést jelent, hogy a könnyen
vizsgálható markerek felhasználhatók a gazdaságilag fontos mennyiségi bélyegek szelekciójában. A markerek és a mennyiségi tulajdonságokat meghatározó szakaszok (QTL-ek) közötti kapcsolatok kiderítéséhez speciális statisztikai elemző programok szükségesek, ezek fejlesztése jelenleg is tart (MÁTYÁS, 2002). A térképezést követő feladat a szakaszokon található gének azonosítása (1. ábra). A mennyiségi tulajdonságot befolyásoló gének a markarekhez való kapcsoltságuk révén mutathatók ki (KOMLÓSI és mtsai, 2000). A genetikai markerek tulajdonságokat meghatározó allélek, amelyeknek sajátosságait fenotípusos, fehérje- vagy DNS-szinten vizsgáljuk. Minél közelebb van a marker a teljesítményt meghatározó génhez (QTL), annál kisebb a rekombináció esélye (KOMLÓSI és mtsai, 2000). A QTL-ek szekvenálásával lehetőség nyílik az összehasonlításra már ismert génekkel, hiszen mint ismeretes, a gének bázissorrendje jelentős mértékben hasonló evolúciósan távoli fajok között is (MÁTYÁS, 2002).
A hagyományos szelekciós eljárásokkal évente 1-3% genetikai előrehaladás érhető el (SMITH, 1984). A hagyományos eljárások alkalmazásának azonban biológiai korlátai is vannak, mint az egyik ivarban mérhető tulajdonság (tej- és tojástermelés, szaporaság) vagy az egy bizonyos korban mérhető tulajdonság (vágási tulajdonságok, életteljesítmény) vagy pedig igen költséges a tulajdonság mérése (betegségre való hajlam). A genetikai markerek viszont bármelyik ivarban és életkorban meghatározhatóak, nem befolyásolja őket a környezet (KOMLÓSI és mtsai, 2000). A gazdasági állatok fontos kvantitatív tulajdonságait (tejhozam, tejzsír- és fehérje mennyiség, hústermelés) különböző gének (QTL) együttes hatása alakítja ki. A tejelő szarvasmarhák esetében számos, a tejösszetételt, illetve a tejhozamot befolyásoló tulajdonságokhoz kapcsolt QTL-t már azonosítottak. Az újabb kutatások ezért elsősorban az egyes QTL-ek markerek segítségével való azonosítására irányulnak. Ezek ismerete és a szelekcióban való felhasználása a genetikai előrehaladást nagymértékben felgyorsíthatja.
A szarvasmarha QTL adatbázisa (Cattle QTL Database) szerint jelenleg (2012-01- 09 adatok szerint, 1. táblázat)), 378 különféle mennyiségi tulajdonsághoz kapcsolt 5207 QTL-t sikerült azonosítani. Ezek közül a legtöbbet, 1485 QTL-t a tejtermeléssel kapcsolatos értékmérő tulajdonságok esetében írtak le. Ha pedig a kromoszómákon való eloszlásukat vesszük figyelembe a legtöbb QTL-t ez idáig a 14-es (469-et) és a 6-os (441- et) kromoszómákon detektálták (www.animalgenome.org) (2-3. melléklet). Mára a kutatások fő célja lett a QTL-ek mögött rejlő mutációkat hordozó gének azonosítása.
1. táblázat: A szarvasmarha fontosabb értékmérő tulajdonság csoportjaiban felfedezett QTL-ek száma (Forrás: www.animalgenom.org).
Fontosabb tulajdonság csoportok QTL-ek száma
Külső / Exterior 123
Egészség / Health 458
Hús / Meat 1091
Tej / Milk 1485
Termelési / Production 1093
Reprodukció / Reproduction 957
6.1.1. A genetikai markerek az állattenyésztésben
Az evolúció során kialakult genetikai variabilitás spontán kereszteződések, gén-, és kromoszómamutációk (pl.: spontán megkettőződés), valamint természetes szelekció eredményeként jött létre. A szelekciót ma a nemesítő tudatosan, saját célkitűzéseinek megfelelően irányítja. A kódó és nem kódoló genomi szakaszok variabilitásának a megismerésével létrejött a genetikai markerek fogalma (JEFFREYS és mtsai, 1985;
WEBER és MAY, 1989). Mint az már az előző fejezetben említésre került a genetikai markerek különböző tulajdonságokat meghatározó változatok (allélek), amelyeknek sajátosságait fenotípusos, fehérje- vagy DNS-szinten vizsgáljuk (HAJÓSNÉ, 1999).
Segítségükkel polimorfizmust, változatosságot keresünk és jellemzünk. Polimorfizmusról akkor beszélünk, ha kettő vagy több eltérő genetikai jelleg egy populációban nagyobb gyakorisággal jelenik meg, mint amelyre az ismétlődő mutáció esetén számíthatunk. Egy lókusz általában akkor polimorf, ha a leggyakoribb allél előfordulása kisebb, mint 0,99.
Az ideális genetikai marker jellemzője hogy, nagymértékben polimorf, allélváltozatait egyértelműen, gyorsan és egyszerűen lehet kimutatni, továbbá előnyös, ha belőlük minél többet sikerül kimutatni a genom bármely területén (DOHY, 1999).
A legrégebbi genetikai markerek látható genetikai jegyek voltak (pl: szőrszín, szarv megléte vagy hiánya), majd a gélelektroforézis technika elterjedésével (60-as évek) a biokémiai (izoenzim) markerek felhasználása vált általánossá. A 80-as évektől terjedtek el a molekuláris markerek, amelyek fehérje és/vagy DNS szinten azonosítják a polimorfizmust.
TANKSLEY és ORTON, (1983) molekuláris markereknek nevezte el azokat a markereket, amelyek fehérje és/vagy DNS-szinten különböztetik meg az egyes alléleket egymástól. A DNS markerek alkalmazásának előnye, hogy nagyfokú variabilitást mutatnak, és a környezet, illetve a génkölcsönhatások nem befolyásolják megjelenésüket.
Előnyük még, hogy, elviekben minden szövetből, az egyed korától, nemétől függetlenül tudunk DNS-t izolálni. Nemcsak élő, hanem élettelen szövetből (pl. többszáz éves csontból) is kinyerhetők, könnyen tárolhatók, és nagyon kis mennyiségekre van szükség. A DNS-szintű markerek alkalmazását a Southern-blot technika, az ezen alapuló RFLP módszer, majd a polimeráz láncreakció (PCR) kidolgozása tette lehetővé. Ezekkel a genetikai vizsgálatok során a bázissorrendben beállt, akár egy bázispárnyi változást is fel lehet használni genetikai markerként. Genomon belüli eloszlásuknak, illetve a magasfokú polimorfizmusuknak köszönhetően egyre jobban előtérbe kerültek a populációgenetikai tanulmányokban (HEDRICK és MILLER, 1992; RUSSEL és mtsai, 1993; VAN ZEVEREN és mtsai, 1995; MAGOULAS, 1998).
A molekuláris genetikai markerek többféle módon is csoportosíthatók. O’BRIEN (1991) két típust határozott meg. Az I. típusú markerek maguk a gének, amelyek kódolt funkcióval rendelkeznek. A II. típusú markerek pedig anonim genetikai szegmensek.
GUPTA és munkatársai (1999) 3 csoportba sorolta a DNS markereket aszerint, hogy milyen vizsgálati elven alapszik a kimutatásuk:
1. hibridizáción alapuló DNS markerek
(első genetációs markerek, melyeket széles körben alkalmaztak a 80-as években)
2. PCR alapú DNS markerek
(a 90-es években voltak alapvető jelentőségűek) 3. DNS chip és szekvenáláson alapuló DNS markerek.
(2000 óta alkalmazott új generációs SNP markerek)
A térképezés is genetikai markerek segítségével történik. A kromoszómán a gének a gyöngysorhoz hasonlóan, lineáris sorrendben, különböző helyeken (lókuszokon) találhatóak. A genetikai térképezésnek két fő célja van. Egyrészt annak a lineáris sorrendnek a meghatározása, ahogy a gének egymáshoz viszonyítva elhelyezkednek, valamint a gének közötti relatív távolság (genetikai távolság) megállapítása. A távolság egysége, annak a kifejezése, hogy a keresztezésben szereplő gének között milyen gyakran jön létre rekombináció. Egy egységnyi térképtávolság (centimorgan) 1%-os rekombinációs gyakoriságnak felel meg.
Összegzésként megállapíthatjuk, hogy a géntérképezési munka egyik fő feladata a kvantitatív (termelési) tulajdonságok kialakulásában fontos szerepet játszó gének, génváltozatok detektálása genetikai markerek segítségével, hogy ezek azonosításával megvalósulhasson a markerek segítségével végezhető szelekció (Marker Assisted Selection - MAS). A markerekre alapozott szelekció (MAS) gyakorlati alkalmazásával a fajták előállítási ideje lerövidíthető, ezáltal költségtakarékosabbá tehető.
A MAS potenciálisan növelheti a genetikai előrehaladás időegységre vonatkoztatott mértékét (DOHY, 1999). Mindezek előfeltétele azonban az adott tulajdonsághoz szorosan kapcsolt markerek detektálása és az állatok genetikai lehetőségeinek hagyományos módon való, magas szintű kiaknázása. A markerek segítségével végezhető szelekció izgalmas kilátásokkal kecsegtet, mert:
várhatóan növeli a tenyészértékbecslés megbízhatóságát,
lehetővé teszi az előszelekció megnyugtató megoldását, ezáltal
csökkenti a generáció-intervallumot, továbbá
fokozhatja a szelekció intenzitását és a nem-additív génhatások (dominancia, szuperdominancia, episztázis) kiaknázhatóságát (DOHY, 1999).
Az eddigi kutatómunkák rengeteg DNS markert tártak fel különböző fajokban, amelyek kisebb-nagyobb mérvű polimorfizmust mutatnak, és alkalmasak egyedek, állományok genotipizálására.
A genotipizálás tehát megteremti a tenyésztők számára a szelekció egészen korai (akár születés után azonnali) lehetőségét. A molekuláris markerekkel különböző számításokat végezhetünk és gyakoriságuk puszta megállapítása mellett további információkat nyerhetünk az állomány homogenitásáról, heterozigozitásáról, a genetikai diverzitásról, valamint a genetikai változatosságról (ZHU és mtsai, 1996a; ACHMANN és mtsai, 2004;
COTHRAN és mtsai, 1997; KOROM és mtsai, 2003; ZSOLNAI és mtsai, 2006). Az állományok összehasonlítására a genetikai távolság (hasonlóság) becslése az alkalmas módszer (NEI, 1972). Mindezek mellett nagy jelentősége van a molekuláris markereknek az állományok, populációk beltenyésztettségi szintjének megállapításában is (CURIK és mtsai, 2003; ZHU és mtsai, 1996b).
6.1.2. Polimorfizmusok, és kimutatásuk eszközei
Egy lókusz egy (monomorf) vagy többféle (polimorf) allélt tartalmazhat. A polimorf lókuszoknak köszönhető a genetikai változatosság. A populáció genetikai változatossága annál nagyobb, minél több gén van több formában jelen. A populáció genetikai heterogenitásának egyik legismertebb jellemzője a polimorf lókuszok %-os aránya. A polimorf lókusz meghatározásához két határértéket találunk a szakirodalomban.
Ezek alapján polimorfnak tekintjük azt a lókuszt, amelyben a leggyakoribb allél gyakorisága kisebb, mint 0,99 ill. 0,95 (HAJÓSNÉ, 1999). Polimorfnak nevezünk minden olyan DNS régiót, lókuszt (mely nem szükségszerűen gén), amelynek különféle szekvencia variánsai léteznek (allélek) a populáció egyes egyedeiben. Abban az esetben, ha valamely kromoszómán a változat (allél) közel helyezkedik el egy adott tulajdonságot meghatározó génhez, vagy az allél maga a géntermék minőségére hat, e polimorf lókusz diagnosztikai jelentőséggel bír (ZSOLNAI, 2000).
A polimorfizmus kifejezés; jelzi, hogy a különbség teljesen semleges hatású is lehet. A betegségokozó változatokat inkább mutációnak szokás nevezni. A mutációk ritkán – kevesebb, mint a populáció 5%-ában – előforduló változatok. A mai kutatási tendencia arra utal, hogy a szekvenciavariációk listája gyorsabban gyűlik, mint ahogy az egyes változatok hatását fel tudnánk mérni. Ezért terjedt el a polimorfizmus elnevezés, jelezve, hogy a szekvenciális különbség hatását legtöbbször nem ismerjük (SASVÁRI-SZÉKELY, 2003). Mint az már az előző fejezetben említésre került, a genetikai variánsok, az allélek, az egyes tulajdonságokat kialakító gének változatai, DNS szekvencia módosulatai. A változás érinthet egyetlen nukleotidot (SNP) vagy ismétlődő szekvenciák ismétlési szám- polimorfizmusában is megnyilvánulhat (2. ábra). A genetikai információ megváltozhat úgy is, hogy egy bázis, vagy akar egy egész szakasz kiesik a DNS láncból (deléció) vagy úgy, hogy új bázisok ékelődnek be (inzerció).
2. ábra: Hosszúságpolimorfizmus és egypontos nukleotid variáció.
Egypontos nukleotid-polimorfizmus (SNP)
Az egypontos nukleotid-polimorfizmus (Single Nucleotide Polymorphism, SNP,
„sznip”) egy DNS szekvencia-variáció, mely akkor jön létre, ha egy nukleotid a genomban megváltozik. Az SNP azt jelenti, hogy a különböző eredetű szekvenciák egy adott pontban többféle variációban létezhetnek. Annak ellenére, hogy a genetikai információ négy betűjéből maximum négyféle eset lehetséges, az SNP-k egy adott helyen legtöbbször csak kétfélék. Például, egy SNP megváltoztathatja az AAGCCTA szekvenciát AAGCTTA szakaszra, de csak akkor tekinthetjük a variációt SNP-nek, ha a populáció legalább 1%- ában megjelenik (3. ábra).
3. ábra: SNP: Egypontos nukleotid-polimorfizmus. Az 1. jelű DNS-szál egy bázispárja különbözik a 2. jelűtől (C/T polimorfizmus). Forrás: http://hu.wikipedia.org/
A sznipek teszik ki a humán genetikai variációk 90%-át, és minden 100-300 bázispáronként megjelenik a humán genomban. Minden 3 SNP-ből 2-ben a citozint timin cseréli le. A SNP-k pontmutációknak tekinthetők, melyek voltak olyan sikeresek az evolúcióban, hogy végül fontos részévé váltak a populációknak.
A mutáció a genom megváltozása, mely pontmutáció vagy kromoszómaaberráció formájában jelentkezik a gyakorlatban. A pontmutáció során egy nukleotidrésznek (bázisnak) egy másik bázissal történő felcserélődéséről van szó. Szerencsés esetben ez nem jelent zavart a fehérjeszintézisben, mivel általában többféle triplet képes ugyanazt az aminosavat kódolni. Gyakoribb, hogy a pontmutáció következtében egy másik aminosav, így másfajta polipeptidlánc szintetizálódik, aminek már hibás vagy csökkent a funkciója, s ez a hibás működés jellegzetes klinikai tünetekben nyilvánul meg (genetikai betegség).
Előfordul, hogy stopkódon (a fehérjeszintézis végét jelző triplet) alakul ki a mutáció következtében, amely csonka, funkcióképtelen fehérje szintéziséhez vezethet. A legtöbb mutáció spontán és váratlanul következik be. Az SNP-k nagy segítséget jelentenek az orvosi kutatásokban, gyógyszerek kifejlesztésében, mivel ezek nem sokat változnak generációról generációra.
Egy bevált módszer az SNP-k kimutatására az RFLP (restriction fragment length polymorphism, azaz restrikciós fragmenthossz-polimorfizmus). Ha egy allél rendelkezik egy restrikciós enzim-hasítóhellyel, míg egy másik nem, akkor a két allél enzimatikus emésztése eltérő hosszúságú fragmenteket fog eredményezni.
Ismétlődő egységek polimorfizmusa
Az egyes egyedek genomjai között található különbségek másik forrása a hosszúságpolimorfizmus, ami egy szekvencia azonos irányultsággal egymás után elhelyezkedő, változó számú ismétlődését jelöli. A genomra általában jellemző, hogy sok benne az ismétlődő szekvenciarészlet. Ez nem csak a „parazita” információra vonatkozik, mert a kódológénekben is előfordulnak ismétlőszakaszok (SASVÁRI-SZÉKELY, 2003).
Egy gén kódoló vagy szabályozó régiójában elhelyezkedő hosszúságpolimorfizmusok hatással lehetnek a kifejeződő fehérjetermék méretére vagy a transzkripció befolyásolásán keresztül mennyiségére.
Ezen polimorfizmusokon kívül még számos, elszórva elhelyezkedő, azonos vagy fordított irányultságú ismétlődő szakasz található (a humán genom 40-50%-át ilyen repetitív szekvenciák alkotják). Nagy számban találhatók például olyan, több ezer bázispár hosszúságú homológ ismétlődő szakaszok, amelyek több gént is magukba foglalhatnak.
Az eltérés az ismétlési számban lehet, ezáltal az adott szakasz hosszabb vagy rövidebb. Az ismétlődő információ egységének hossza változó. Vannak nagyon rövid ismétlések (short tandem repeats, STR), ahol 1–5 betűből álló egység ismétlődik egymás után sokszor. Más esetben ez egy hosszabb információs egység. Az ilyen hosszabb ismétléseket változó számú egymás utáni ismétlése a VNTR (Variable Number of Tandem Repeats).
A molekuláris genetikai elemzések során a kétféle enzimtechnikát (restrikciós és polimeráz) kombinálni is lehet. Az eljárásokat aszerint szokás csoportosítani, hogy PCR- alapúak-e vagy nem, továbbá, hogy véletlenszerűen amplifikáló primert használnak-e, vagy meghatározott célszakaszokat vizsgálnak (ZSOLNAI, 2000).
A továbbiakban a polimorfizmusokat kimutató eljárásokat az azonosításuk szempontjából történő osztályozásuk alapján mutatom be.
A polimorfizmusok detektálása alapulhat:
a különböző allélok eltérő fizikai tulajdonságainak észlelésén (a DNS szál olvadási hőmérséklete, másodlagos szerkezet megváltozása vagy eltérő hossz kialakulása),
az allélok sokszorosítási reakcióban mutatott, a primerrel közösen meghatározott láncnövekedési esélyének megváltozásán,
metiláció érzékeny vagy metilációra érzéketlen enzimekkel szemben mutatott viselkedésen,
az egyszálú szekvencia változatok egymással szembeni affinitás különbségein.
Az alábbi összefoglaló táblázatban (2. táblázat) szereplő eljárások, módszerek a leírt detektálási alaptípusok valamelyikét, vagy azok kombinációját alkalmazzák.
2. táblázat: Polimorfizmusok kimutatásához használt eljárások
Vizsgálati módszerek, eljárások Rövid leírás Több genomhely (multilókusz) vizsgálat RFLP
(Restriction Fragment Length Polymorphism = restrikciós fragmenthossz polimorfizmus)
Egy RFLP mintázat használható kapcsoltságok meghatározására (BOTSTEIN és mtsai, 1980), vagy fragmentek izolálására további szekvencia vizsgálatokhoz.
„Ujjlenyomat generálása” PCR- rel
RAPD (Random
Amplified Polimorphic
DNA = random
amplifikált polimorf DNS)
DAF (DNA
Amplification
Fingerprinting = DNS amplifikációs
ujjlenyomat)
AP-PCR (Arbitrary Primered PCR = PCR tetszés szerinti primerrel)
A vizsgálatok célja reprodukálható genetikai ujjlenyomat (fingerprint) elérése pl. egy keresett tulajdonsággal kapcsolt DNS régió megtalálása érdekében (BASSAM és BENTLEY, 1994).
AFLP (Amplified Fragment Length Polymorphism = amplifikált fragmenthossz polimorfizmus)
A DNS restrikciós enzimekkel történő emésztése után 20-30 bázispárból álló adapter szekvenciákat kapcsolnak a fragmentekhez, majd PCR segítségével a fragmentumokat szelektíven felszaporítják. Jellemzője, hogy nagyszámú mintázatot ad, viszont a többi markerezési technikához képest a fragmentumok kicsik.
Egyedi genomhely vizsgálat Mikroszatellitek
STR (Short Tandem Repeat = rövid tandem ismétlődés)
SSR (Simple Sequence Repeat = egyszerű szekvencia ismétlődés)
A miroszatellitek kiváló eszközei a géntérképezésnek, valamint használatukkal egyedek könnyen és pontosan azonosíthatóak.
Tipizálásuk annak a néhány bázispár ismétlődésének számbeli változékonyságán alapul, melyek két, csak egy-egy adott genomrészletre jellemző szekvenciarészlet között helyezkedik el (WEBER és MAY, 1989).
ASA (Allele Specific Amplification = allél specifikus amplifikáció)
További elnevezések lehetnek:
ASP (Allele Specific PCR), ARMS (Amplification Refractory Mutation System), PASA (PCR Amplification of Specific Alleles)
A módszer pontmutációk detektálására alkalmas a PCR termékeinek utólagos manipulációja nélkül.
PCR-RFLP A legáltalánosabban használt módszer ismert mutációk azonosítására.
LCR (Ligase Chain Reaction = ligáz láncreakció)
A módszer lényege, hogy a hőstabil ligáz és több oligonukleotid alkalmazásával pontmutáció detektálható (WIEDMANN és mtsai, 1994).
SSCP (Single-Strand Conformation Polymorphism = egyszálú DNS konfirmációs polimorfizmus)
Az SSCP analízis azon az elven alapszik, hogy a vad típusú DNS szálhoz képest pontmutációt, inzerciót vagy deléciót tartalmazó mutáns DNS egyszálú állapotban más másodlagos szerkezetet vesz fel, ami által az elválasztó mátrixban a mozgékonysága is más lesz.
Használatával a mutációk 70-95%-a detektálható (NATARAJ és mtsai, 1999).
Egyedi lókusz egymás melletti, sokaságban való kimutatása
Szekvenálás A szekvenálás a DNS lánc bázissorendjének meghatározását és a mutációk felderítését szolgálja.
Mikroszekvenálás A variábilis nukleotid körüli régiót amplifikálják és a kapott PCR terméket szilárd hordozóhoz kötik, ahol az egyszálú DNS-ként van jelen. A reakciókomponensek eltávolítása után a szilárd hordozón kapott, beépült nukleotidon lévő fluoreszcens festék jele alapján a genotípus azonosítható.
Piroszekvenálás A piroszekvenálás olyan valós idejű módszer, amellyel a láncépítés során felszabaduló inorganikus pirofoszfát csoport mennyiségével arányos emittált fény nagyságából következtetnek a szekvenciára (NORDSTRÖM és mtsai, 2000).
Chipek használata A szilárd felülethez kötött egyszálú DNS vagy DNS-t imitáló molekula bázisszekvenciája a felületen elfoglalt pozíciójával együtt ismert. A megismerni kívánt szekvencia egynukleotidos polimorfizmus környezetének kiegészítő szekvenciáját szisztematikusan variálva és sorba állítva várjuk a feldarabolt és jelzett genomiális minta vagy PCR-rel sokszorosított, a próbákkal hasonló hosszúságú termékek erős hibridizációját a megfelelő bázisvariációval rendelkező próbához.
Következő generációs szekvenálási technológiák (NGS-Next Generation Sequencing)
Az ezredfordulót követően a DNS-könyvtárakból klonálisan amplifikált DNS-molekulákat különböző módszerek segítségével szekvenáló (454 Life Science, Illumina és Applied Biosystems) következő generációs szekvenálási technológiák jelentek meg. Ezekben közös, hogy sok szálon párhuzamosan folyik a DNS szekvenálása (MIHÁLY és GYŐRFFY, 2011).
Life Technologies
Ion Torrent szekvencia analízis
A technológia a DNS-molekulát félvezetők segítségével dekódolja, ezáltal a DNS-szekvenálás folyamata lényegesen felgyorsítható. Az Ion Torrent elnevezésű berendezésnek mindössze két órára van szüksége a DNS szekvenálásához, noha a minta előkészítése ennél tovább tart. Az új eszköz az első kereskedelmi rendszer, amely közvetlenül egy félvezető chipen fejti meg a DNS betűsorát, és amely feszültségváltozást mér fény helyett. Mintegy 1,2 millió miniatűr üreget maratnak bele egy chip felszínébe, és megtöltik a meghatározandó DNS- darabokat tartalmazó "gyöngysorokkal". Az üregek alján lévő detektor érzékeli az üregekben lévő oldat savasságát, amely minden egyes újabb DNS-szálnak a gyöngysorhoz való kapcsolódásakor megváltozik. A néhány másodperces ciklust addig ismétlik, amíg a DNS-lánc minden egyes bázisegységét azonosítják (MTI Sajtóadatbank, 2011).
454 Life Science A módszerrel 500 millió bázisnyi nyers szekvencia képezhető le néhány óra alatt. Az egyszálú DNS-t egy gyöngyre kötik, és
A továbbiakban a vizsgálataim során használt valós idejű polimeráz láncreakció, valamint a PCR-RFLP módszereket mutatom be részletesebben.
Roche 454 Sequencing:
a „sequencing by syntesis”
módszerét alkalmazzák
ezután egy méhkaptárszerű lapra helyezik, amelyen apró lyukak vannak, ahová egyszerre csak egyetlen gyöngy fér be. Itt piroszekvenálás segítségével olvassák le a szekvenciát (MARGULIES és mtsai, 2005). A reakció során a polimeráz meghosszabbítja a DNS-szálat a platekhez ciklikusan hozzáadott fluoreszcens jelet adó nukleotidokkal (RONAGHI és mtsai, 1998). Végül a ciklus végen lemossák a fluoreszcens nukleotidot, és a következő ciklusban egy újabb nukleotidot épít be a polimeráz. A sikeres nukleotidbekötődést fotonemisszió jelzi, amit egy CCD kamera detektál. Végül az adatfeldolgozást követően válnak megismerhetővé a szekvenciák (www.454.com).
Illumina:
a „pair-end” szekvenálási módszert alkalmazzák
Először DNS-könyvtárakat hoznak létre úgynevezett híd- amplifikáció révén (KORBEL és mtsai 2007). Az egyszálú DNS darabok végére kötött adaptervégződés segítségével oligonukleotid horgonyokhoz hibridizálva immobilizálják a DNS-fragmentumokat. A lekötött egyszálú DNS másik szálát primerek segítségével megszintetizálják és a szabad végükre kötött adaptervegződéssel hídszerűen meghajlítva kihorgonyozzák a nukleotidszálakat. Az amplifikációt követően a reverz szálakat eltávolítják és a csoportokban klonálisan amplifikált nukleotidszálakon végzik a szekvenálást fluoreszcensen jelölt nukleotidok segítségével. Minden egyes nukleotid kötődése után detektáljak a fluoreszcens jelet, végül lemossák a festéket, mielőtt egy újabb ciklusba kezdenek (www.illumina.com).
Applied Biosystems
Az ABI „mate-paired library” a teljesgenom-szekvenálás mellett még célzott reszekvenálásra,
génexpresszió mérésre és kromatin- immunoprecipitációs szekvenálásra (ChIP-Seq) is alkalmazható.
A DNS-könyvtárak tartalmát klonálisan amplifikálják gyöngyökre, majd PCR-reakcióval megsokszorozzák, és a gyöngyöket kovalens kötéssel egy tárgylemezhez kötik. A szekvenálást két ismert bázisból álló próbákkal végzik, így a 4 különböző fluoreszcens festékkel minden kötés első és második bázisa megismerhető. A komplementerpróbák hibridizálódnak a leolvasandó szekvenciához, majd végül a fluoreszcens jel mérése révén határozzák meg a szekvenciát (www.appliedbiosystems.com).
Második generációs szekvenálási technológiák
Az utobbi 1-2 évben jelent meg a következő generációs szekvenálási technológiák második generációja, mint például a Helicos, Pacific Biosciences, Nanopore vagy a NABsys szekvenátorai. Ezek a cégek az amplifikációs lépést átugorva „single-molecule sequencing”
technológiákat alkalmazva az egyes molekulákat hatarozzák meg (MIHÁLY és GYŐRFFY, 2011).